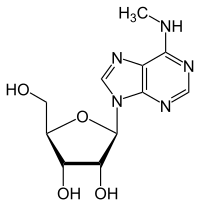
N6-Methyladenosin
| Strukturformel | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||
| Allgemeines | |||||||||||||
| Name | N6-Methyladenosin | ||||||||||||
| Andere Namen |
| ||||||||||||
| Summenformel | C11H15N5O4 | ||||||||||||
| Kurzbeschreibung |
weißer Feststoff[1] | ||||||||||||
| Externe Identifikatoren/Datenbanken | |||||||||||||
| |||||||||||||
| Eigenschaften | |||||||||||||
| Molare Masse | 281,27 g·mol−1 | ||||||||||||
| Aggregatzustand |
fest[1] | ||||||||||||
| Schmelzpunkt |
>147 °C (Zersetzung)[1] | ||||||||||||
| Sicherheitshinweise | |||||||||||||
| |||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen (0 °C, 1000 hPa). | |||||||||||||
N6-Methyladenosin (m6A) wurde erstmals in den 1970er Jahren identifiziert und charakterisiert[3][4][5][6] und ist eine häufige Modifikation in mRNA und DNA.[7] Es kommt in einigen Viren[5][6][8][9] und den meisten Eukaryoten einschließlich Säugetieren,[3][4][10][11] Insekten,[12] Pflanzen[13][14][15] und Hefe[16][17] vor. Sie findet sich auch in tRNA, rRNA und small nuclear RNA (snRNA) sowie in einigen langen nicht-kodierenden RNAs wie Xist.[18][19]
Die Methylierung von Adenosin wird durch einen großen m6A-Methyltransferase-Komplex gesteuert, der METTL3 enthält, die Untereinheit, die S-Adenosyl-L-Methionin (SAM) bindet.[20] In vitro methyliert dieser Methyltransferase-Komplex bevorzugt RNA-Oligonukleotide, die GGACU enthalten,[21] und eine ähnliche Präferenz wurde in vivo für die m6A-Stellen in der genomischen RNA des Rous-Sarkom-Virus[22] und in der bovinen Prolaktin-mRNA[23] beobachtet. In neueren Studien wurden weitere Schlüsselkomponenten des m6A-Methyltransferase-Komplexes in Säugetieren charakterisiert, darunter METTL14,[24][25] Wilms Tumor 1 assoziiertes Protein (WTAP),[24][26] VIRMA[27] und METTL5.[28] Nach Spekulationen im Jahr 2010, dass m6A in der mRNA dynamisch und reversibel ist,[29] bestätigte die Entdeckung der ersten m6A-Demethylase, dem Fettmasse- und Adipositas-assoziierten Protein (FTO), im Jahr 2011 diese Hypothese[30]. Eine zweite m6A-Demethylase, alkB homolog 5 (ALKBH5), wurde später entdeckt.[31]
Die biologischen Funktionen von m6A werden durch eine Gruppe von RNA-bindenden Proteinen vermittelt, die spezifisch methyliertes Adenosin auf RNA erkennen. Diese Bindungsproteine werden als m6A-Reader bezeichnet. Die Proteine der YT521-B-Homologie (YTH)-Domänenfamilie (YTHDF1, YTHDF2, YTHDF3 und YTHDC1) wurden als direkte m6A-Reader charakterisiert und besitzen eine konservierte m6A-Bindungstasche.[19][32][33][34][35] Insulinähnlicher Wachstumsfaktor-2 mRNA-bindende Proteine 1 (IGF2BP1), 2 (IGF2BP2), and 3 (IGF2BP3) wurden als eine neue Klasse von m6A-Reader beschrieben.[36] IGF2BPs nutzen K-Homologie (KH)-Domänen, um selektiv m6A-haltige RNAs zu erkennen und deren Translation und Stabilität zu fördern.[36] Diese m6A-Reader bilden zusammen mit m6A-Methyltransferasen (Writers) und Demethylasen (Erasers) einen komplexen Mechanismus der m6A-Regulation, in dem Writers und Erasers die Verteilung von m6Aauf der RNA bestimmen, während die Reader m6A-abhängige Funktionen vermitteln. m6A vermittelt außerdem einen strukturellen Schalter, den sogenannten m6A-Switch.[37]
Die Spezifität der m6A-Installation auf der mRNA wird durch die Exon-Architektur und Exon-Junction-Komplexe (EJC) kontrolliert. Exon-Junction-Komplexe unterdrücken die m6A-Methylierung in der Nähe von Exon-Exon-Grenzen, indem sie die RNA in ihrer Nähe verpacken und vor der Methylierung durch den m6A-Methyltransferase-Komplex schützen. m6A-Regionen in langen internen und terminalen Exons, die von Exon-Exon-Grenzen und Exon-Junction-Komplexen entfernt sind, entgehen der Unterdrückung durch Exon-Junction-Komplexe und können von dem Methyltransferase-Komplex methyliert werden.[38]
Hintergrund
N6-Methyladenosin (m6A) ist ein Nukleosid und kommt in der tRNA, rRNA, mRNA, ncRNA und snRNA vor.[39] Es besteht aus der β-D-Ribofuranose (Zucker) und dem N6-Methyladenin, ist ein Derivat des Adenosins, welches an der Aminogruppe methyliert ist und entsteht enzymatisch durch Methylierung von Adenosin mittels der N6-Adenosin-Methyltransferase.
Die dimethylierte Variante ist das N6,N6-Dimethyladenosin.
Vorkommen in Arten
Hefe
In der Knospenhefe (Saccharomyces cerevisiae) wird die Expression des Homologs von METTL3, IME4, in diploiden Zellen als Reaktion auf einen Mangel an Stickstoff und fermentierbaren Kohlenstoffquellen induziert und ist für die mRNA-Methylierung und die fehlerlose Initiierung der Meiose und Sporulation notwendig.[16][17] mRNAs von IME1 und IME2 (frühe Regulatoren der Meiose), sind als Ziele der Methylierung bekannt, ebenso wie die Transkripte von IME4 selbst.[17]
Pflanzen
In Pflanzen liegt der größte Teil von m6A innerhalb von 150 Nukleotiden vor dem Beginn des Poly(A)-Schwanzes.[40]
Mutationen in MTA, dem Homologen von METTL3 in Arabidopsis thaliana, führen zum Stillstand des Embryos im Kugelstadium. Eine >90%ige Reduktion des m6A-Spiegels in reifen Pflanzen führt zu dramatisch veränderten Wachstumsmustern und homöotischen Anomalien der Blüten.[40]
Säugetiere
Die Kartierung von m6A in der RNA von Mensch und Maus ergab mehr als 18.000 m6A-Stellen in den Transkripten von mehr als 7.000 menschlichen Genen mit der Konsensussequenz [G/A/U][G>A]m6AC[U>A/C],[18][19][41] die mit dem zuvor identifizierten Motiv übereinstimmt. Die Lokalisation einzelner m6A-Stellen in vielen mRNAs ist bei Mensch und Maus sehr ähnlich,[18][19] und transkriptomweite Analysen zeigen, dass m6A in Regionen mit hoher evolutionärer Konservierung zu finden ist.[18] m6A findet sich innerhalb langer interner Exons und ist bevorzugt in 3'-UTRs und um Stoppcodons angereichert. m6A innerhalb von 3'-UTRs ist auch mit dem Vorhandensein von microRNA-Bindungsstellen assoziiert; etwa 2/3 der mRNAs, die eine m6A-Stelle innerhalb ihres 3'-UTRs enthalten, besitzen auch mindestens eine microRNA-Bindungsstelle.[18] Durch die Integration von m6A-Sequenzierungsdaten hat die Datenbank RMBase ~200.000 Stellen in den Genomen von Mensch und Maus identifiziert und verfügbar gemacht, die N6-Methyladenosinen (m6A) in RNA entsprechen.[41]
Die genaue Kartierung von m6A durch m6A-CLIP/IP (kurz m6A-CLIP) ergab, dass sich der Großteil von m6A im letzten Exon von mRNAs in verschiedenen Geweben/kultivierten Zellen von Maus und Mensch befindet, und dass die m6A-Anreicherung um Stoppcodons nicht signifikant ist, da sich viele Stoppcodons um den Anfang der letzten Exons liegen, wo m6A tatsächlich angereichert vorkommt.[42] Das hohe Vorkommen von m6A im letzten Exon (>=70 %) deutet auf eine mögliche Regulation der 3'UTR einschließlich alternativer Polyadenylierung hin.[42] Die Kombination von m6A-CLIP mit strenger Zellfraktionierungsbiochemie zeigt, dass m6A-mRNA-Modifikationen in naszierende pre-mRNAs eingebaut werden und nicht für das Spleißen notwendig sind, aber den zytoplasmatischen Umsatz spezifizieren.[43][44]
m6A unterliegt einer dynamischen Regulation sowohl während der Entwicklung als auch als Antwort auf zelluläre Stimuli. Die Analyse von m6A in der RNA des Gehirns von Mäusen zeigt, dass die m6A-Konzentrationen während der Embryonalentwicklung niedrig sind und im Erwachsenenalter drastisch ansteigen.[18] In mESCs und während der Entwicklung von Mäusen konnte gezeigt werden, dass FTO die Demethylierung von m6A in der LINE1-RNA vermittelt und dadurch den lokalen Chromatinstatus und die Transkription benachbarter Gene beeinflusst.[45] Darüber hinaus hat das Silencing der m6A-Methyltransferase signifikante Auswirkungen auf die Genexpression und alternative RNA-Spleißmuster, was zu einer Modulation des p53-Signalwegs (auch bekannt als TP53) und zu Apoptose führt.[19]
m6A findet sich auch auf den RNA-Komponenten der R-Schleifen in menschlichen Zellen, wo es an der Regulation der Stabilität von RNA:DNA-Hybriden beteiligt ist.[46]
Die Bedeutung der m6A-Methylierung für physiologische Prozesse wurde kürzlich nachgewiesen. Die Hemmung der m6A-Methylierung durch pharmakologische Inhibition der zellulären Methylierung oder spezifischer durch siRNA-vermitteltes Silencing der m6A-Methylase METTL3 führte zu einer Verlängerung der circadianen Periode. Im Gegensatz dazu führte eine Überexpression von METTL3 zu einer Verkürzung der Periode. Die zirkadiane Uhr der Säugetiere, die aus einer transkriptionellen Rückkopplungsschleife besteht, die so reguliert ist, dass sie mit einer Periode von etwa 24 Stunden oszilliert, reagiert also sehr empfindlich auf Störungen der m6A-abhängigen RNA-Prozessierung, was wahrscheinlich auf die Anwesenheit von m6A-Stellen in den Gentranskripten der Uhr zurückzuführen ist.[47][48] Die Auswirkungen einer globalen Inhibition der Methylierung auf die circadiane Periode in Mauszellen können durch die ektopische Expression eines Enzyms aus dem bakteriellen Methylmetabolismus verhindert werden. Mauszellen, die dieses bakterielle Protein exprimieren, sind resistent gegen eine pharmakologische Hemmung des Methylmetabolismus und zeigen keine Abnahme der mRNA-Methylierung oder der Proteinmethylierung.[49]
Klinische Relevanz
Angesichts der vielfältigen Funktionen von m6A in verschiedenen physiologischen Prozessen ist es nicht verwunderlich, dass Verbindungen zwischen m6A und zahlreichen menschlichen Krankheiten gefunden wurden, von denen viele auf Mutationen oder Einzelnukleotid-Polymorphismen (SNPs) verwandter Faktoren von m6A zurückzuführen sind. Zusammenhänge zwischen m6A und zahlreichen Krebsarten wurden bei Magenkrebs, Prostatakrebs, Brustkrebs, Bauchspeicheldrüsenkrebs, Nierenkrebs, Mesotheliom, Sarkom und Leukämie nachgewiesen.[50][51][52][53][54][55][56][57][58][59][60][61] Eventuell stärkere Auswirkungen von m6A auf die Proliferation von Krebszellen sind Gegenstand laufender Forschung. Bereits bekannt ist, dass die Depletion von METTL3 die Apoptose von Krebszellen induziert und die Invasivität von Krebszellen reduziert,[62][63] während die Aktivierung von ALKBH5 durch Hypoxie nachweislich zur Akkumulation von Krebsstammzellen führt.[64] m6A wurde auch für die Regulation der Energiehomöostase und Fettleibigkeit in Betracht gezogen, da FTO ein wichtiges Regulatorgen des Energiestoffwechsels ist. Es wurde gezeigt, dass SNPs von FTO mit dem Body-Mass-Index in menschlichen Populationen und dem Auftreten von Adipositas und Diabetes assoziiert sind.[65][66][67][68][69] Vermutlich beeinflusst FTO auch die Differenzierung von Adipozyten.[70][71][72] Der Zusammenhang zwischen m6A und neuronalen Störungen wurde ebenfalls untersucht. Beispielsweise könnten neurodegenerative Erkrankungen durch m6A beeinflusst werden, da die dopaminerge kognitive Signalübertragung von FTO und der korrekten m6A-Methylierung wichtiger Signaltranskripte abhängt.[73] Es ist bekannt, dass Mutationen in HNRNPA2B1, einem potentiellen Leser von m6A, Neurodegeneration verursachen können.[74] IGF2BP1-3, eine neue Klasse von m6A-Lesern, hat onkogene Funktionen. Der Knockdown oder Knockout von IGF2BP1-3 reduzierte die Expression des MYC-Proteins, die Zellproliferation und die Koloniebildung in menschlichen Krebszelllinien.[36] ZC3H13, ein Protein des m6A-Methyltransferase-Komplexes, hemmt bei Knockout signifikant das Wachstum von Darmkrebszellen.[75]
Außerdem hat m6A einen Einfluss auf Virusinfektionen. Es ist bekannt, dass viele RNA-Viren wie SV40, Adenoviren, Herpesviren, Rous-Sarkom-Viren und Influenzaviren eine interne m6A-Methylierung auf der genomischen RNA des Virus aufweisen.[76] Mehrere Studien konnten zeigen, dass m6A-Regulatoren die Effizienz der Infektion, Replikation, Translation und des Transports von RNA-Viren wie dem humanen Immundefizienz-Virus (HIV), dem Hepatitis-B-Virus (HBV), dem Hepatitis-C-Virus (HCV) und dem Zika-Virus (ZIKV) kontrollieren.[77][78][79][80][81][82] Diese Ergebnisse deuten darauf hin, dass m6A und seine verwandten Faktoren eine entscheidende Rolle bei der Regulation der viralen Lebenszyklen und der Wirt-Virus-Interaktionen spielen.
Neben den Auswirkungen auf die Viren selbst können m6A-Modifikationen auch die angeborene Immunantwort stören. So konnte bei HBV gezeigt werden, dass m6A-Modifikationen die Erkennung von Viren durch RIG-1, einen Mustererkennungsrezeptor des Immunsystems, stören. Modifikationen können auch nachgeschaltete Signalwege über Mechanismen wie Ubiquitinierung und Veränderungen der Proteinexpression stören.[82]
In Bakterien
Die m6A-Methylierung ist auch in Bakterien weit verbreitet und beeinflusst Funktionen wie DNA-Replikation, -Reparatur und -Genexpression sowie die prokaryotische Abwehr.
Bei der Replikation markieren m6A-Modifikationen DNA-Regionen, in denen die Initiationsphase stattfindet, und regulieren über die Dam-Methyltransferase in E. coli den zeitlichen Ablauf.[83][84] Ein weiteres Enzym, die Dam-DNA-Methylase, reguliert die Mismatch-Reparatur mit Hilfe von m6A-Modifikationen, die andere Reparaturproteine beeinflussen, indem sie spezifische Mismatches erkennen.[85]
In einigen Fällen des DNA-Schutzes spielen m6A-Methylierungen (zusammen mit m4C-Modifikationen) eine Rolle beim Schutz bakterieller DNA, indem sie bestimmte Endonukleasen über das Restriktionsmodifikationssystem beeinflussen und so den Einfluss von Bakteriophagen verringern. Eine solche Rolle besteht in der Einführung einer Methyltransferase, die die gleiche Zielstelle erkennt, die von Restriktionsenzymen (Typ-1-Restriktionsenzyme) angegriffen wird, und in der Modifizierung dieser Enzyme, um sie daran zu hindern, die bakterielle DNA anzugreifen.[86][87]
Im Entwicklungsstadium
Es konnte nachgewiesen werden, dass m6A-Modifikationen in Verbindung mit anderen epigenetischen Veränderungen eine wesentliche Rolle während der eukaryontischen Entwicklung spielen. Diesbezüglich wurden m6A-Modifikationen bei hämatopoetischen Stammzellen (HSCs), neuronalen Stammzellen (NSCs) sowie primordialen Keimzellen (PCGs) während des Wachstums und der Differenzierung festgestellt. Je nach Entwicklungsstadium können Veränderungen an HSCs die Stammzelldifferenzierung entweder fördern oder hemmen, indem sie den Übergang von Epithelzellen zu hämopoetischen Zellen durch Hemmung oder Verminderung von METTL3 beeinflussen. Modifikationen an neuronalen Stammzellen (NSCs) können Veränderungen der Gehirngröße, der Neuronenbildung, des Langzeitgedächtnisses und der Lernfähigkeit bewirken. Diese Veränderungen werden häufig durch die Hemmung von METTL- oder YTHDF-readers und -writers verursacht. Im Reproduktionssystem stören m6A-Veränderungen nachweislich den Übergang von mütterlicher zu zygotischer mRNA und wirken sich negativ auf die Bildung von Gameten und die Fruchtbarkeit aus. Ähnlich wie bei NSCs ist die Hemmung der Proteinfamilien METTL und YTHDF häufig ein Katalysator für diese Veränderungen.[88]
Weblinks
- Eintrag zu N6-Methyladenosine in der Human Metabolome Database (HMDB), abgerufen am 12. Oktober 2013.
- Modification Summary von N6-Methyladenosine in der Modomics-Datenbank, abgerufen am 13. Januar 2014.
Einzelnachweise
- ↑ a b c Eintrag zu N6-Methyladenosine bei Toronto Research Chemicals, abgerufen am 23. Januar 2022 (PDF).
- ↑ Dieser Stoff wurde in Bezug auf seine Gefährlichkeit entweder noch nicht eingestuft oder eine verlässliche und zitierfähige Quelle hierzu wurde noch nicht gefunden.
- ↑ a b Jerry M. Adams, Suzanne Cory: Modified nucleosides and bizarre 5′-termini in mouse myeloma mRNA. In: Nature. Band 255, Nr. 5503, 1. Mai 1975, S. 28–33, doi:10.1038/255028a0.
- ↑ a b Ronald Desrosiers, Karen Friderici, Fritz Rottman: Identification of Methylated Nucleosides in Messenger RNA from Novikoff Hepatoma Cells. In: Proceedings of the National Academy of Sciences. Band 71, Nr. 10, Oktober 1974, S. 3971–3975, doi:10.1073/pnas.71.10.3971.
- ↑ a b Y Aloni, R Dhar, G Khoury: Methylation of nuclear simian virus 40 RNAs. In: Journal of Virology. Band 32, Nr. 1, Oktober 1979, S. 52–60, doi:10.1128/jvi.32.1.52-60.1979.
- ↑ a b Karen Beemon, Jerry Keith: Localization of N6-methyladenosine in the Rous sarcoma virus genome. In: Journal of Molecular Biology. Band 113, Nr. 1, Juni 1977, S. 165–179, doi:10.1016/0022-2836(77)90047-X.
- ↑ Pengfei Ji, Xia Wang, Nina Xie, Yujing Li: N6-Methyladenosine in RNA and DNA: An Epitranscriptomic and Epigenetic Player Implicated in Determination of Stem Cell Fate. In: Stem Cells International. Band 2018, 10. Oktober 2018, S. 1–18, doi:10.1155/2018/3256524.
- ↑ David G. Courtney, Edward M. Kennedy, Rebekah E. Dumm, Hal P. Bogerd, Kevin Tsai, Nicholas S. Heaton, Bryan R. Cullen: Epitranscriptomic Enhancement of Influenza A Virus Gene Expression and Replication. In: Cell Host & Microbe. Band 22, Nr. 3, September 2017, S. 377–386.e5, doi:10.1016/j.chom.2017.08.004.
- ↑ Nandan S. Gokhale, Alexa B.R. McIntyre, Michael J. McFadden, Allison E. Roder, Edward M. Kennedy, Jorge A. Gandara, Sharon E. Hopcraft, Kendra M. Quicke, Christine Vazquez, Jason Willer, Olga R. Ilkayeva, Brittany A. Law, Christopher L. Holley, Mariano A. Garcia-Blanco, Matthew J. Evans, Mehul S. Suthar, Shelton S. Bradrick, Christopher E. Mason, Stacy M. Horner: N6 -Methyladenosine in Flaviviridae Viral RNA Genomes Regulates Infection. In: Cell Host & Microbe. Band 20, Nr. 5, November 2016, S. 654–665, doi:10.1016/j.chom.2016.09.015.
- ↑ Cha Mer Wei, Alan Gershowitz, Bernard Moss: 5'-Terminal and internal methylated nucleotide sequences in HeLa cell mRNA. In: Biochemistry. Band 15, Nr. 2, 1. Januar 1976, S. 397–401, doi:10.1021/bi00647a024.
- ↑ Robert P. Perry, Dawn E. Kelley, Karen Friderici, Fritz Rottman: The methylated constituents of L cell messenger RNA: Evidence for an unusual cluster at the 5′ terminus. In: Cell. Band 4, Nr. 4, April 1975, S. 387–394, doi:10.1016/0092-8674(75)90159-2.
- ↑ Robbet Levis, Sheldon Penman: 5′-Terminal structures of poly(A)+ cytoplasmic messenger RNA and of poly(A)+ and poly(A)− heterogeneous nuclear RNA of cells of the dipteran Drosophila melanogaster. In: Journal of Molecular Biology. Band 120, Nr. 4, April 1978, S. 487–515, doi:10.1016/0022-2836(78)90350-9.
- ↑ J.L. Nichols: in maize poly(A)-containing RNA. In: Plant Science Letters. Band 15, Nr. 4, August 1979, S. 357–361, doi:10.1016/0304-4211(79)90141-X.
- ↑ T. D. Kennedy, B. G. Lane: Wheat embryo ribonucleates. XIII. Methyl-substituted nucleoside constituents and 5′-terminal dinucleotide sequences in bulk poly(A)-rich RNA from imbibing wheat embryos. In: Canadian Journal of Biochemistry. Band 57, Nr. 6, 1. Juni 1979, S. 927–931, doi:10.1139/o79-112.
- ↑ Silin Zhong, Hongying Li, Zsuzsanna Bodi, James Button, Laurent Vespa, Michel Herzog, Rupert G. Fray: MTA Is an Arabidopsis Messenger RNA Adenosine Methylase and Interacts with a Homolog of a Sex-Specific Splicing Factor. In: The Plant Cell. Band 20, Nr. 5, 30. Juni 2008, S. 1278–1288, doi:10.1105/tpc.108.058883.
- ↑ a b M. J. Clancy: Induction of sporulation in Saccharomyces cerevisiae leads to the formation of N6-methyladenosine in mRNA: a potential mechanism for the activity of the IME4 gene. In: Nucleic Acids Research. Band 30, Nr. 20, 15. Oktober 2002, S. 4509–4518, doi:10.1093/nar/gkf573.
- ↑ a b c Zsuzsanna Bodi, James D. Button, Donald Grierson, Rupert G. Fray: Yeast targets for mRNA methylation. In: Nucleic Acids Research. Band 38, Nr. 16, September 2010, S. 5327–5335, doi:10.1093/nar/gkq266.
- ↑ a b c d e f Kate D. Meyer, Yogesh Saletore, Paul Zumbo, Olivier Elemento, Christopher E. Mason, Samie R. Jaffrey: Comprehensive Analysis of mRNA Methylation Reveals Enrichment in 3′ UTRs and near Stop Codons. In: Cell. Band 149, Nr. 7, Juni 2012, S. 1635–1646, doi:10.1016/j.cell.2012.05.003.
- ↑ a b c d e Dan Dominissini, Sharon Moshitch-Moshkovitz, Schraga Schwartz, Mali Salmon-Divon, Lior Ungar, Sivan Osenberg, Karen Cesarkas, Jasmine Jacob-Hirsch, Ninette Amariglio, Martin Kupiec, Rotem Sorek, Gideon Rechavi: Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. In: Nature. Band 485, Nr. 7397, 10. Mai 2012, S. 201–206, doi:10.1038/nature11112.
- ↑ J.A. Bokar, M.E. Rath-Shambaugh, R. Ludwiczak, P. Narayan, F. Rottman: Characterization and partial purification of mRNA N6-adenosine methyltransferase from HeLa cell nuclei. Internal mRNA methylation requires a multisubunit complex. In: Journal of Biological Chemistry. Band 269, Nr. 26, Juli 1994, S. 17697–17704, doi:10.1016/s0021-9258(17)32497-3.
- ↑ Joan E. Harper, Sheila M. Miceli, Richard J. Roberts, James L. Manley: Sequence specificity of the human mRNA N6-adenosine methylase in vitro. In: Nucleic Acids Research. Band 18, Nr. 19, 1990, S. 5735–5741, doi:10.1093/nar/18.19.5735.
- ↑ S E Kane, K Beemon: Precise localization of m6A in Rous sarcoma virus RNA reveals clustering of methylation sites: implications for RNA processing. In: Molecular and Cellular Biology. Band 5, Nr. 9, September 1985, S. 2298–2306, doi:10.1128/MCB.5.9.2298.
- ↑ S Horowitz, A Horowitz, T W Nilsen, T W Munns, F M Rottman: Mapping of N6-methyladenosine residues in bovine prolactin mRNA. In: Proceedings of the National Academy of Sciences. Band 81, Nr. 18, September 1984, S. 5667–5671, doi:10.1073/pnas.81.18.5667.
- ↑ a b Jianzhao Liu, Yanan Yue, Dali Han, Xiao Wang, Ye Fu, Liang Zhang, Guifang Jia, Miao Yu, Zhike Lu, Xin Deng, Qing Dai, Weizhong Chen, Chuan He: A METTL3–METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. In: Nature Chemical Biology. Band 10, Nr. 2, Februar 2014, S. 93–95, doi:10.1038/nchembio.1432.
- ↑ Yang Wang, Yue Li, Julia I. Toth, Matthew D. Petroski, Zhaolei Zhang, Jing Crystal Zhao: N6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells. In: Nature Cell Biology. Band 16, Nr. 2, Februar 2014, S. 191–198, doi:10.1038/ncb2902.
- ↑ Xiao-Li Ping, Bao-Fa Sun, Lu Wang, Wen Xiao, Xin Yang, Wen-Jia Wang, Samir Adhikari, Yue Shi, Ying Lv, Yu-Sheng Chen, Xu Zhao, Ang Li, Ying Yang, Ujwal Dahal, Xiao-Min Lou, Xi Liu, Jun Huang, Wei-Ping Yuan, Xiao-Fan Zhu, Tao Cheng, Yong-Liang Zhao, Xinquan Wang, Jannie M Rendtlew Danielsen, Feng Liu, Yun-Gui Yang: Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase. In: Cell Research. Band 24, Nr. 2, Februar 2014, S. 177–189, doi:10.1038/cr.2014.3.
- ↑ Schraga Schwartz, Maxwell R. Mumbach, Marko Jovanovic, Tim Wang, Karolina Maciag, G. Guy Bushkin, Philipp Mertins, Dmitry Ter-Ovanesyan, Naomi Habib, Davide Cacchiarelli, Neville E. Sanjana, Elizaveta Freinkman, Michael E. Pacold, Rahul Satija, Tarjei S. Mikkelsen, Nir Hacohen, Feng Zhang, Steven A. Carr, Eric S. Lander, Aviv Regev: Perturbation of m6A Writers Reveals Two Distinct Classes of mRNA Methylation at Internal and 5′ Sites. In: Cell Reports. Band 8, Nr. 1, Juli 2014, S. 284–296, doi:10.1016/j.celrep.2014.05.048.
- ↑ Nhan van Tran, Felix G M Ernst, Ben R Hawley, Christiane Zorbas, Nathalie Ulryck, Philipp Hackert, Katherine E Bohnsack, Markus T Bohnsack, Samie R Jaffrey, Marc Graille, Denis L J Lafontaine: The human 18S rRNA m6A methyltransferase METTL5 is stabilized by TRMT112. In: Nucleic Acids Research. Band 47, Nr. 15, 5. September 2019, S. 7719–7733, doi:10.1093/nar/gkz619.
- ↑ Chuan He: Grand Challenge Commentary: RNA epigenetics? In: Nature Chemical Biology. Band 6, Nr. 12, Dezember 2010, S. 863–865, doi:10.1038/nchembio.482.
- ↑ Guifang Jia, Ye Fu, Xu Zhao, Qing Dai, Guanqun Zheng, Ying Yang, Chengqi Yi, Tomas Lindahl, Tao Pan, Yun-Gui Yang, Chuan He: N6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. In: Nature Chemical Biology. Band 7, Nr. 12, Dezember 2011, S. 885–887, doi:10.1038/nchembio.687.
- ↑ Guanqun Zheng, John Arne Dahl, Yamei Niu, Peter Fedorcsak, Chun-Min Huang, Charles J. Li, Cathrine B. Vågbø, Yue Shi, Wen-Ling Wang, Shu-Hui Song, Zhike Lu, Ralph P.G. Bosmans, Qing Dai, Ya-Juan Hao, Xin Yang, Wen-Ming Zhao, Wei-Min Tong, Xiu-Jie Wang, Florian Bogdan, Kari Furu, Ye Fu, Guifang Jia, Xu Zhao, Jun Liu, Hans E. Krokan, Arne Klungland, Yun-Gui Yang, Chuan He: ALKBH5 Is a Mammalian RNA Demethylase that Impacts RNA Metabolism and Mouse Fertility. In: Molecular Cell. Band 49, Nr. 1, Januar 2013, S. 18–29, doi:10.1016/j.molcel.2012.10.015.
- ↑ Xiao Wang, Zhike Lu, Adrian Gomez, Gary C. Hon, Yanan Yue, Dali Han, Ye Fu, Marc Parisien, Qing Dai, Guifang Jia, Bing Ren, Tao Pan, Chuan He: N6-methyladenosine-dependent regulation of messenger RNA stability. In: Nature. Band 505, Nr. 7481, Januar 2014, S. 117–120, doi:10.1038/nature12730.
- ↑ Xiao Wang, Boxuan Simen Zhao, Ian A. Roundtree, Zhike Lu, Dali Han, Honghui Ma, Xiaocheng Weng, Kai Chen, Hailing Shi, Chuan He: N6-methyladenosine Modulates Messenger RNA Translation Efficiency. In: Cell. Band 161, Nr. 6, Juni 2015, S. 1388–1399, doi:10.1016/j.cell.2015.05.014.
- ↑ Chao Xu, Xiao Wang, Ke Liu, Ian A Roundtree, Wolfram Tempel, Yanjun Li, Zhike Lu, Chuan He, Jinrong Min: Structural basis for selective binding of m6A RNA by the YTHDC1 YTH domain. In: Nature Chemical Biology. Band 10, Nr. 11, November 2014, S. 927–929, doi:10.1038/nchembio.1654.
- ↑ Wen Xiao, Samir Adhikari, Ujwal Dahal, Yu-Sheng Chen, Ya-Juan Hao, Bao-Fa Sun, Hui-Ying Sun, Ang Li, Xiao-Li Ping, Wei-Yi Lai, Xing Wang, Hai-Li Ma, Chun-Min Huang, Ying Yang, Niu Huang, Gui-Bin Jiang, Hai-Lin Wang, Qi Zhou, Xiu-Jie Wang, Yong-Liang Zhao, Yun-Gui Yang: Nuclear m 6 A Reader YTHDC1 Regulates mRNA Splicing. In: Molecular Cell. Band 61, Nr. 4, Februar 2016, S. 507–519, doi:10.1016/j.molcel.2016.01.012.
- ↑ a b c Huilin Huang, Hengyou Weng, Wenju Sun, Xi Qin, Hailing Shi, Huizhe Wu, Boxuan Simen Zhao, Ana Mesquita, Chang Liu, Celvie L. Yuan, Yueh-Chiang Hu, Stefan Hüttelmaier, Jennifer R. Skibbe, Rui Su, Xiaolan Deng, Lei Dong, Miao Sun, Chenying Li, Sigrid Nachtergaele, Yungui Wang, Chao Hu, Kyle Ferchen, Kenneth D. Greis, Xi Jiang, Minjie Wei, Lianghu Qu, Jun-Lin Guan, Chuan He, Jianhua Yang, Jianjun Chen: Recognition of RNA N6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation. In: Nature Cell Biology. Band 20, Nr. 3, März 2018, S. 285–295, doi:10.1038/s41556-018-0045-z.
- ↑ Nian Liu, Qing Dai, Guanqun Zheng, Chuan He, Marc Parisien, Tao Pan: N6-methyladenosine-dependent RNA structural switches regulate RNA–protein interactions. In: Nature. Band 518, Nr. 7540, 26. Februar 2015, S. 560–564, doi:10.1038/nature14234.
- ↑ P. Cody He, Jiangbo Wei, Xiaoyang Dou, Bryan T. Harada, Zijie Zhang, Ruiqi Ge, Chang Liu, Li-Sheng Zhang, Xianbin Yu, Shuai Wang, Ruitu Lyu, Zhongyu Zou, Mengjie Chen, Chuan He: Exon architecture controls mRNA m 6 A suppression and gene expression. In: Science. Band 379, Nr. 6633, 17. Februar 2023, S. 677–682, doi:10.1126/science.abj9090.
- ↑ Patrick A. Limbach, Pamela F. Crain, James A. McCloskey: Summary: the modified nucleosides of RNA. In: Nucleic Acids Research, 22, 1994. Nummer 12, S. 2183–2196 doi:10.1093/nar/22.12.2183, PMID 7518580, PMC 523672 (freier Volltext).
- ↑ a b Zsuzsanna Bodi, Silin Zhong, Surbhi Mehra, Jie Song, Neil Graham, Hongying Li, Sean May, Rupert George Fray: Adenosine Methylation in Arabidopsis mRNA is Associated with the 3′ End and Reduced Levels Cause Developmental Defects. In: Frontiers in Plant Science. Band 3, 2012, doi:10.3389/fpls.2012.00048.
- ↑ a b Wen-Ju Sun, Jun-Hao Li, Shun Liu, Jie Wu, Hui Zhou, Liang-Hu Qu, Jian-Hua Yang: RMBase: a resource for decoding the landscape of RNA modifications from high-throughput sequencing data. In: Nucleic Acids Research. Band 44, D1, 4. Januar 2016, S. D259–D265, doi:10.1093/nar/gkv1036.
- ↑ a b Shengdong Ke, Endalkachew A. Alemu, Claudia Mertens, Emily Conn Gantman, John J. Fak, Aldo Mele, Bhagwattie Haripal, Ilana Zucker-Scharff, Michael J. Moore, Christopher Y. Park, Cathrine Broberg Vågbø, Anna Kusśnierczyk, Arne Klungland, James E. Darnell, Robert B. Darnell: A majority of m 6 A residues are in the last exons, allowing the potential for 3′ UTR regulation. In: Genes & Development. Band 29, Nr. 19, 1. Oktober 2015, S. 2037–2053, doi:10.1101/gad.269415.115.
- ↑ Shengdong Ke, Amy Pandya-Jones, Yuhki Saito, John J. Fak, Cathrine Broberg Vågbø, Shay Geula, Jacob H. Hanna, Douglas L. Black, James E. Darnell, Robert B. Darnell: m 6 A mRNA modifications are deposited in nascent pre-mRNA and are not required for splicing but do specify cytoplasmic turnover. In: Genes & Development. Band 31, Nr. 10, 15. Mai 2017, S. 990–1006, doi:10.1101/gad.301036.117.
- ↑ Nicolle A. Rosa-Mercado, Johanna B. Withers, Joan A. Steitz: Settling the m 6 A debate: methylation of mature mRNA is not dynamic but accelerates turnover. In: Genes & Development. Band 31, Nr. 10, 15. Mai 2017, S. 957–958, doi:10.1101/gad.302695.117.
- ↑ Jiangbo Wei, Xianbin Yu, Lei Yang, Xuelian Liu, Boyang Gao, Boxian Huang, Xiaoyang Dou, Jun Liu, Zhongyu Zou, Xiao-Long Cui, Li-Sheng Zhang, Xingsen Zhao, Qinzhe Liu, P. Cody He, Caraline Sepich-Poore, Nicole Zhong, Wenqiang Liu, Yanhe Li, Xiaochen Kou, Yanhong Zhao, You Wu, Xuejun Cheng, Chuan Chen, Yiming An, Xueyang Dong, Huanyu Wang, Qiang Shu, Ziyang Hao, Tao Duan, Yu-Ying He, Xuekun Li, Shaorong Gao, Yawei Gao, Chuan He: FTO mediates LINE1 m 6 A demethylation and chromatin regulation in mESCs and mouse development. In: Science. Band 376, Nr. 6596, 27. Mai 2022, S. 968–973, doi:10.1126/science.abe9582.
- ↑ Abdulkadir Abakir, Tom C. Giles, Agnese Cristini, Jeremy M. Foster, Nan Dai, Marta Starczak, Alejandro Rubio-Roldan, Miaomiao Li, Maria Eleftheriou, James Crutchley, Luke Flatt, Lorraine Young, Daniel J. Gaffney, Chris Denning, Bjørn Dalhus, Richard D. Emes, Daniel Gackowski, Ivan R. Corrêa, Jose L. Garcia-Perez, Arne Klungland, Natalia Gromak, Alexey Ruzov: N6-methyladenosine regulates the stability of RNA:DNA hybrids in human cells. In: Nature Genetics. Band 52, Nr. 1, Januar 2020, S. 48–55, doi:10.1038/s41588-019-0549-x.
- ↑ Jean-Michel Fustin, Masao Doi, Yoshiaki Yamaguchi, Hayashi Hida, Shinichi Nishimura, Minoru Yoshida, Takayuki Isagawa, Masaki Suimye Morioka, Hideaki Kakeya, Ichiro Manabe, Hitoshi Okamura: RNA-Methylation-Dependent RNA Processing Controls the Speed of the Circadian Clock. In: Cell. Band 155, Nr. 4, November 2013, S. 793–806, doi:10.1016/j.cell.2013.10.026.
- ↑ Michael H. Hastings: m6A mRNA Methylation: A New Circadian Pacesetter. In: Cell. Band 155, Nr. 4, November 2013, S. 740–741, doi:10.1016/j.cell.2013.10.028.
- ↑ Jean-Michel Fustin, Shiqi Ye, Christin Rakers, Kensuke Kaneko, Kazuki Fukumoto, Mayu Yamano, Marijke Versteven, Ellen Grünewald, Samantha J. Cargill, T. Katherine Tamai, Yao Xu, Maria Luísa Jabbur, Rika Kojima, Melisa L. Lamberti, Kumiko Yoshioka-Kobayashi, David Whitmore, Stephanie Tammam, P. Lynne Howell, Ryoichiro Kageyama, Takuya Matsuo, Ralf Stanewsky, Diego A. Golombek, Carl Hirschie Johnson, Hideaki Kakeya, Gerben van Ooijen, Hitoshi Okamura: Methylation deficiency disrupts biological rhythms from bacteria to humans. In: Communications Biology. Band 3, Nr. 1, 6. Mai 2020, doi:10.1038/s42003-020-0942-0.
- ↑ Ainur R. Akilzhanova, Bagdat Nyshanbekkyzy, Zhannur M. Nurkina, Ivan I. Shtephanov, Abay K. Makishev, Tasbolat A. Adylkhanov, Tolebay K. Rakhypbekov, Erlan M. Ramanculov, Kuvat T. Momynaliev: BRCA1 and BRCA2 Gene Mutations Screening In Sporadic Breast Cancer Patients In Kazakhstan. In: Central Asian Journal of Global Health. Band 2, Nr. 1, 21. Mai 2013, doi:10.5195/cajgh.2013.29.
- ↑ S M Reddy, M Sadim, J Li, N Yi, S Agarwal, C S Mantzoros, V G Kaklamani: Clinical and genetic predictors of weight gain in patients diagnosed with breast cancer. In: British Journal of Cancer. Band 109, Nr. 4, August 2013, S. 872–881, doi:10.1038/bjc.2013.441.
- ↑ Katrin-Janine Heiliger, Julia Hess, Donata Vitagliano, Paolo Salerno, Herbert Braselmann, Giuliana Salvatore, Clara Ugolini, Isolde Summerer, Tatjana Bogdanova, Kristian Unger, Gerry Thomas, Massimo Santoro, Horst Zitzelsberger: Novel candidate genes of thyroid tumourigenesis identified in Trk-T1 transgenic mice. In: Endocrine-Related Cancer. Band 19, Nr. 3, Juni 2012, S. 409–421, doi:10.1530/ERC-11-0387.
- ↑ Angeles Ortega, Martina Niksic, Angela Bachi, Matthias Wilm, Lucas Sánchez, Nicholas Hastie, Juan Valcárcel: Biochemical Function of Female-Lethal (2)D/Wilms' Tumor Suppressor-1-associated Proteins in Alternative Pre-mRNA Splicing. In: Journal of Biological Chemistry. Band 278, Nr. 5, Januar 2003, S. 3040–3047, doi:10.1074/jbc.M210737200.
- ↑ Du‐Il Jin, Sang Weon Lee, Myoung‐Eun Han, Hyun‐Jung Kim, Seon‐Ae Seo, Gi‐Yeong Hur, Shin Jung, Bong‐Seon Kim, Sae‐Ock Oh: Expression and roles of W ilms' tumor 1‐associating protein in glioblastoma. In: Cancer Science. Band 103, Nr. 12, Dezember 2012, S. 2102–2109, doi:10.1111/cas.12022.
- ↑ Yingsong Lin, Junko Ueda, Kiyoko Yagyu, Hiroshi Ishii, Makoto Ueno, Naoto Egawa, Haruhisa Nakao, Mitsuru Mori, Keitaro Matsuo, Shogo Kikuchi: Association between variations in the fat mass and obesity-associated gene and pancreatic cancer risk: a case–control study in Japan. In: BMC Cancer. Band 13, Nr. 1, Dezember 2013, doi:10.1186/1471-2407-13-337.
- ↑ R. Casalegno-Garduño, A. Schmitt, X. Wang, X. Xu, M. Schmitt: Wilms' Tumor 1 as a Novel Target for Immunotherapy of Leukemia. In: Transplantation Proceedings. Band 42, Nr. 8, Oktober 2010, S. 3309–3311, doi:10.1016/j.transproceed.2010.07.034.
- ↑ Michael Linnebacher, Anne Wienck, Inga Boeck, Ernst Klar: Identification of an MSI-H Tumor-Specific Cytotoxic T Cell Epitope Generated by the ( − 1) Frame of U79260(FTO). In: Journal of Biomedicine and Biotechnology. Band 2010, 2010, S. 1–6, doi:10.1155/2010/841451.
- ↑ M. J. Machiela, S. Lindstrom, N. E. Allen, C. A. Haiman, D. Albanes, A. Barricarte, S. I. Berndt, H. B. Bueno-de-Mesquita, S. Chanock, J. M. Gaziano, S. M. Gapstur, E. Giovannucci, B. E. Henderson, E. J. Jacobs, L. N. Kolonel, V. Krogh, J. Ma, M. J. Stampfer, V. L. Stevens, D. O. Stram, A. Tjonneland, R. Travis, W. C. Willett, D. J. Hunter, L. Le Marchand, P. Kraft: Association of Type 2 Diabetes Susceptibility Variants With Advanced Prostate Cancer Risk in the Breast and Prostate Cancer Cohort Consortium. In: American Journal of Epidemiology. Band 176, Nr. 12, 15. Dezember 2012, S. 1121–1129, doi:10.1093/aje/kws191.
- ↑ Jirong Long, Ben Zhang, Lisa B. Signorello, Qiuyin Cai, Sandra Deming-Halverson, Martha J. Shrubsole, Maureen Sanderson, Joe Dennis, Kyriaki Michailiou, Douglas F. Easton, Xiao-Ou Shu, William J. Blot, Wei Zheng: Evaluating Genome-Wide Association Study-Identified Breast Cancer Risk Variants in African-American Women. In: PLoS ONE. Band 8, Nr. 4, 8. April 2013, S. e58350, doi:10.1371/journal.pone.0058350.
- ↑ Virginia Kaklamani, Nengjun Yi, Maureen Sadim, Kalliopi Siziopikou, Kui Zhang, Yanfei Xu, Sarah Tofilon, Surbhi Agarwal, Boris Pasche, Christos Mantzoros: The role of the fat mass and obesity associated gene (FTO) in breast cancer risk. In: BMC Medical Genetics. Band 12, Nr. 1, Dezember 2011, doi:10.1186/1471-2350-12-52.
- ↑ Brandon L. Pierce, Melissa A. Austin, Habibul Ahsan: Association study of type 2 diabetes genetic susceptibility variants and risk of pancreatic cancer: an analysis of PanScan-I data. In: Cancer Causes & Control. Band 22, Nr. 6, Juni 2011, S. 877–883, doi:10.1007/s10552-011-9760-5.
- ↑ Joseph A. Bokar: The biosynthesis and functional roles of methylated nucleosides in eukaryotic mRNA. In: Fine-Tuning of RNA Functions by Modification and Editing. Band 12. Springer Berlin Heidelberg, Berlin, Heidelberg 2005, ISBN 978-3-540-24495-0, S. 141–177, doi:10.1007/b106365.
- ↑ Shuibin Lin, Junho Choe, Peng Du, Robinson Triboulet, Richard I. Gregory: The m 6 A Methyltransferase METTL3 Promotes Translation in Human Cancer Cells. In: Molecular Cell. Band 62, Nr. 3, Mai 2016, S. 335–345, doi:10.1016/j.molcel.2016.03.021.
- ↑ Chuanzhao Zhang, Debangshu Samanta, Haiquan Lu, John W. Bullen, Huimin Zhang, Ivan Chen, Xiaoshun He, Gregg L. Semenza: Hypoxia induces the breast cancer stem cell phenotype by HIF-dependent and ALKBH5-mediated m 6 A-demethylation of NANOG mRNA. In: Proceedings of the National Academy of Sciences. Band 113, Nr. 14, 5. April 2016, doi:10.1073/pnas.1602883113.
- ↑ Ruth J. F. Loos, Giles S. H. Yeo: The bigger picture of FTO—the first GWAS-identified obesity gene. In: Nature Reviews Endocrinology. Band 10, Nr. 1, Januar 2014, S. 51–61, doi:10.1038/nrendo.2013.227.
- ↑ Timothy M. Frayling, Nicholas J. Timpson, Michael N. Weedon, Eleftheria Zeggini, Rachel M. Freathy, Cecilia M. Lindgren, John R. B. Perry, Katherine S. Elliott, Hana Lango, Nigel W. Rayner, Beverley Shields, Lorna W. Harries, Jeffrey C. Barrett, Sian Ellard, Christopher J. Groves, Bridget Knight, Ann-Marie Patch, Andrew R. Ness, Shah Ebrahim, Debbie A. Lawlor, Susan M. Ring, Yoav Ben-Shlomo, Marjo-Riitta Jarvelin, Ulla Sovio, Amanda J. Bennett, David Melzer, Luigi Ferrucci, Ruth J. F. Loos, IneÌs Barroso, Nicholas J. Wareham, Fredrik Karpe, Katharine R. Owen, Lon R. Cardon, Mark Walker, Graham A. Hitman, Colin N. A. Palmer, Alex S. F. Doney, Andrew D. Morris, George Davey Smith, Andrew T. Hattersley, Mark I. McCarthy: A Common Variant in the FTO Gene Is Associated with Body Mass Index and Predisposes to Childhood and Adult Obesity. In: Science. Band 316, Nr. 5826, 11. Mai 2007, S. 889–894, doi:10.1126/science.1141634.
- ↑ Lina Wang, Qing Yu, Yan Xiong, Linfei Liu, Xuening Zhang, Zhen Zhang, Jianru Wu, Bei Wang: Variant rs1421085 in the FTO gene contribute childhood obesity in Chinese children aged 3–6years. In: Obesity Research & Clinical Practice. Band 7, Nr. 1, Januar 2013, S. e14–e22, doi:10.1016/j.orcp.2011.12.007.
- ↑ Ineta Kalnina, Linda Zaharenko, Iveta Vaivade, Vita Rovite, Liene Nikitina-Zake, Raitis Peculis, Davids Fridmanis, Kristine Geldnere, Josefin A. Jacobsson, Markus S. Almen, Valdis Pirags, Helgi B. Schiöth, Janis Klovins: Polymorphisms in FTO and near TMEM18 associate with type 2 diabetes and predispose to younger age at diagnosis of diabetes. In: Gene. Band 527, Nr. 2, September 2013, S. 462–468, doi:10.1016/j.gene.2013.06.079.
- ↑ Efthimia Karra, Owen G. O’Daly, Agharul I. Choudhury, Ahmed Yousseif, Steven Millership, Marianne T. Neary, William R. Scott, Keval Chandarana, Sean Manning, Martin E. Hess, Hiroshi Iwakura, Takashi Akamizu, Queensta Millet, Cigdem Gelegen, Megan E. Drew, Sofia Rahman, Julian J. Emmanuel, Steven C.R. Williams, Ulrich U. Rüther, Jens C. Brüning, Dominic J. Withers, Fernando O. Zelaya, Rachel L. Batterham: A link between FTO, ghrelin, and impaired brain food-cue responsivity. In: Journal of Clinical Investigation. Band 123, Nr. 8, 1. August 2013, S. 3539–3551, doi:10.1172/JCI44403.
- ↑ Xu Zhao, Ying Yang, Bao-Fa Sun, Yue Shi, Xin Yang, Wen Xiao, Ya-Juan Hao, Xiao-Li Ping, Yu-Sheng Chen, Wen-Jia Wang, Kang-Xuan Jin, Xing Wang, Chun-Min Huang, Yu Fu, Xiao-Meng Ge, Shu-Hui Song, Hyun Seok Jeong, Hiroyuki Yanagisawa, Yamei Niu, Gui-Fang Jia, Wei Wu, Wei-Min Tong, Akimitsu Okamoto, Chuan He, Jannie M Rendtlew Danielsen, Xiu-Jie Wang, Yun-Gui Yang: FTO-dependent demethylation of N6-methyladenosine regulates mRNA splicing and is required for adipogenesis. In: Cell Research. Band 24, Nr. 12, Dezember 2014, S. 1403–1419, doi:10.1038/cr.2014.151.
- ↑ Myrte Merkestein, Samantha Laber, Fiona McMurray, Daniel Andrew, Gregor Sachse, Jeremy Sanderson, Mengdi Li, Samuel Usher, Dyan Sellayah, Frances M. Ashcroft, Roger D. Cox: FTO influences adipogenesis by regulating mitotic clonal expansion. In: Nature Communications. Band 6, Nr. 1, 17. April 2015, doi:10.1038/ncomms7792.
- ↑ Meizi Zhang, Ying Zhang, Jun Ma, Feima Guo, Qian Cao, Yu Zhang, Bin Zhou, Jijie Chai, Wenqing Zhao, Renbin Zhao: The Demethylase Activity of FTO (Fat Mass and Obesity Associated Protein) Is Required for Preadipocyte Differentiation. In: PLOS ONE. Band 10, Nr. 7, 28. Juli 2015, S. e0133788, doi:10.1371/journal.pone.0133788.
- ↑ Martin E Hess, Simon Hess, Kate D Meyer, Linda A W Verhagen, Linda Koch, Hella S Brönneke, Marcelo O Dietrich, Sabine D Jordan, Yogesh Saletore, Olivier Elemento, Bengt F Belgardt, Thomas Franz, Tamas L Horvath, Ulrich Rüther, Samie R Jaffrey, Peter Kloppenburg, Jens C Brüning: The fat mass and obesity associated gene (Fto) regulates activity of the dopaminergic midbrain circuitry. In: Nature Neuroscience. Band 16, Nr. 8, August 2013, S. 1042–1048, doi:10.1038/nn.3449.
- ↑ Hong Joo Kim, Nam Chul Kim, Yong-Dong Wang, Emily A. Scarborough, Jennifer Moore, Zamia Diaz, Kyle S. MacLea, Brian Freibaum, Songqing Li, Amandine Molliex, Anderson P. Kanagaraj, Robert Carter, Kevin B. Boylan, Aleksandra M. Wojtas, Rosa Rademakers, Jack L. Pinkus, Steven A. Greenberg, John Q. Trojanowski, Bryan J. Traynor, Bradley N. Smith, Simon Topp, Athina-Soragia Gkazi, Jack Miller, Christopher E. Shaw, Michael Kottlors, Janbernd Kirschner, Alan Pestronk, Yun R. Li, Alice Flynn Ford, Aaron D. Gitler, Michael Benatar, Oliver D. King, Virginia E. Kimonis, Eric D. Ross, Conrad C. Weihl, James Shorter, J. Paul Taylor: Mutations in prion-like domains in hnRNPA2B1 and hnRNPA1 cause multisystem proteinopathy and ALS. In: Nature. Band 495, Nr. 7442, März 2013, S. 467–473, doi:10.1038/nature11922.
- ↑ Ze-Lin Wang, Bin Li, Yu-Xia Luo, Qiao Lin, Shu-Rong Liu, Xiao-Qin Zhang, Hui Zhou, Jian-Hua Yang, Liang-Hu Qu: Comprehensive Genomic Characterization of RNA-Binding Proteins across Human Cancers. In: Cell Reports. Band 22, Nr. 1, Januar 2018, S. 286–298, doi:10.1016/j.celrep.2017.12.035.
- ↑ Prema Narayan, Fritz M. Rottman: Methylation of mRNA. In: Advances in Enzymology - and Related Areas of Molecular Biology. 1. Auflage. Band 65. Wiley, 1992, ISBN 978-0-471-52760-2, S. 255–285, doi:10.1002/9780470123119.ch7.
- ↑ Edward M. Kennedy, Hal P. Bogerd, Anand V.R. Kornepati, Dong Kang, Delta Ghoshal, Joy B. Marshall, Brigid C. Poling, Kevin Tsai, Nandan S. Gokhale, Stacy M. Horner, Bryan R. Cullen: Posttranscriptional m 6 A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. In: Cell Host & Microbe. Band 19, Nr. 5, Mai 2016, S. 675–685, doi:10.1016/j.chom.2016.04.002.
- ↑ Nagaraja Tirumuru, Boxuan Simen Zhao, Wuxun Lu, Zhike Lu, Chuan He, Li Wu: N6-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. In: eLife. Band 5, 2. Juli 2016, doi:10.7554/eLife.15528.
- ↑ Gianluigi Lichinchi, Shang Gao, Yogesh Saletore, Gwendolyn Michelle Gonzalez, Vikas Bansal, Yinsheng Wang, Christopher E. Mason, Tariq M. Rana: Dynamics of the human and viral m6A RNA methylomes during HIV-1 infection of T cells. In: Nature Microbiology. Band 1, Nr. 4, 22. Februar 2016, doi:10.1038/nmicrobiol.2016.11.
- ↑ Gianluigi Lichinchi, Boxuan Simen Zhao, Yinga Wu, Zhike Lu, Yue Qin, Chuan He, Tariq M. Rana: Dynamics of Human and Viral RNA Methylation during Zika Virus Infection. In: Cell Host & Microbe. Band 20, Nr. 5, November 2016, S. 666–673, doi:10.1016/j.chom.2016.10.002.
- ↑ Nandan S. Gokhale, Alexa B.R. McIntyre, Michael J. McFadden, Allison E. Roder, Edward M. Kennedy, Jorge A. Gandara, Sharon E. Hopcraft, Kendra M. Quicke, Christine Vazquez, Jason Willer, Olga R. Ilkayeva, Brittany A. Law, Christopher L. Holley, Mariano A. Garcia-Blanco, Matthew J. Evans, Mehul S. Suthar, Shelton S. Bradrick, Christopher E. Mason, Stacy M. Horner: N6 -Methyladenosine in Flaviviridae Viral RNA Genomes Regulates Infection. In: Cell Host & Microbe. Band 20, Nr. 5, November 2016, S. 654–665, doi:10.1016/j.chom.2016.09.015.
- ↑ a b Jae-Su Moon, Wooseong Lee, Yong-Hee Cho, Yonghyo Kim, Geon-Woo Kim: The Significance of N6-Methyladenosine RNA Methylation in Regulating the Hepatitis B Virus Life Cycle. In: Journal of Microbiology and Biotechnology. Band 34, Nr. 2, 28. März 2024, S. 233–239, doi:10.4014/jmb.2309.09013.
- ↑ Zach Klapholz O’Brown, Eric Lieberman Greer: N6-Methyladenine: A Conserved and Dynamic DNA Mark. In: DNA Methyltransferases - Role and Function. Band 945. Springer International Publishing, Cham 2016, ISBN 978-3-319-43622-7, S. 213–246, doi:10.1007/978-3-319-43624-1_10.
- ↑ Melania Balzarolo, Sander Engels, Anja J. de Jong, Katka Franke, Timo K. van den Berg, Muhammet F. Gulen, Andrea Ablasser, Edith M. Janssen, Bas van Steensel, Monika C. Wolkers: m6A methylation potentiates cytosolic dsDNA recognition in a sequence-specific manner. In: Open Biology. Band 11, Nr. 3, März 2021, doi:10.1098/rsob.210030.
- ↑ Nalini Raghunathan, Sayantan Goswami, Jakku K Leela, Apuratha Pandiyan, Jayaraman Gowrishankar: A new role for Escherichia coli Dam DNA methylase in prevention of aberrant chromosomal replication. In: Nucleic Acids Research. Band 47, Nr. 11, 20. Juni 2019, S. 5698–5711, doi:10.1093/nar/gkz242.
- ↑ Matthew J. Blow, Tyson A. Clark, Chris G. Daum, Adam M. Deutschbauer, Alexey Fomenkov, Roxanne Fries, Jeff Froula, Dongwan D. Kang, Rex R. Malmstrom, Richard D. Morgan, Janos Posfai, Kanwar Singh, Axel Visel, Kelly Wetmore, Zhiying Zhao, Edward M. Rubin, Jonas Korlach, Len A. Pennacchio, Richard J. Roberts: The Epigenomic Landscape of Prokaryotes. In: PLOS Genetics. Band 12, Nr. 2, 12. Februar 2016, S. e1005854, doi:10.1371/journal.pgen.1005854.
- ↑ W. A. M. Loenen, D. T. F. Dryden, E. A. Raleigh, G. G. Wilson: Type I restriction enzymes and their relatives. In: Nucleic Acids Research. Band 42, Nr. 1, 1. Januar 2014, S. 20–44, doi:10.1093/nar/gkt847.
- ↑ Xiulin Jiang, Baiyang Liu, Zhi Nie, Lincan Duan, Qiuxia Xiong, Zhixian Jin, Cuiping Yang, Yongbin Chen: The role of m6A modification in the biological functions and diseases. In: Signal Transduction and Targeted Therapy. Band 6, Nr. 1, 21. Februar 2021, doi:10.1038/s41392-020-00450-x.




