Kombinierte Malon- und Methylmalonazidurie
Kombinierte Malon und Methylmalonazidurie, auch CMAMMA oder kombinierte Malon- und Methylmalonazidämie genannt, ist eine angeborene Stoffwechselkrankheit, die durch die biochemische Beobachtung von erhöhten Mengen von Malonsäure und Methylmalonsäure gekennzeichnet ist, wobei die Methylmalonsäurewerte höher sind als die Malonsäurewerte.[1][2] CMAMMA ist nicht nur eine Organoazidopathie, sondern auch ein Defekt der mitochondrialen Fettsäuresynthese (mtFASII).[3] Forscher sagen voraus, dass CMAMMA eine der häufigsten Formen der Methylmalonazidurie und vielleicht eine der häufigsten angeborenen Stoffwechselstörungen ist, die jedoch aufgrund des Hindurchrutschens bei diagnostischen Verfahren oft unentdeckt bleibt.[4][5]
Symptome und Anzeichen
Die klinischen Phänotypen der CMAMMA sind sehr heterogen und reichen von asymptomatischen, über leichte bis hin zu schweren Symptomen.[6][7] Die dahinterliegende Pathophysiologie ist noch nicht verstanden.[3] In der Literatur werden folgende Symptome genannt:
- Psychische Erkrankungen[4]
- Gedächtnisprobleme[4]
- kognitiver Abbau[4]
Wenn die ersten Symptome in der Kindheit auftreten, handelt es sich eher um intermediäre Stoffwechselstörungen, während es sich bei Erwachsenen meist um neurologische Symptome handelt.[4][6]
Ursachen
CMAMMA ist eine angeborene, autosomal-rezessive Stoffwechselstörung, mit der Folge eines Mangels des Enzyms ACSF3. Das ACSF3-Gen ist auf Chromosom 16 Genlocus q24.3 verortet. Es besteht aus 11 Exons und kodiert ein aus 576 Aminosäuren bestehendes Protein.[6][7] CMAMMA kann durch homozygote oder auch compound-heterozygote Varianten im Gen ACSF3 verursacht sein. Ausgehend von der Frequenz des selteneren Alleles (MAF) ergibt sich für CMAMMA eine Bevölkerungsinzidenz von ca. 1:30 000.[4]
Pathophysiologie
Das Gen ACSF3 kodiert eine Acyl-CoA-Synthetase, welche in den Mitochondrien lokalisiert ist und eine hohe Spezifität für Malonsäure und Methylmalonsäure aufweist.[9] Es ist als Malonyl-CoA-Synthetase für die Umwandlung von Malonsäure in Malonyl-CoA und als Methylmalonyl-CoA-Synthetase für die Umwandlung von Methylmalonsäure in Methylmalonyl-CoA verantwortlich.[10]
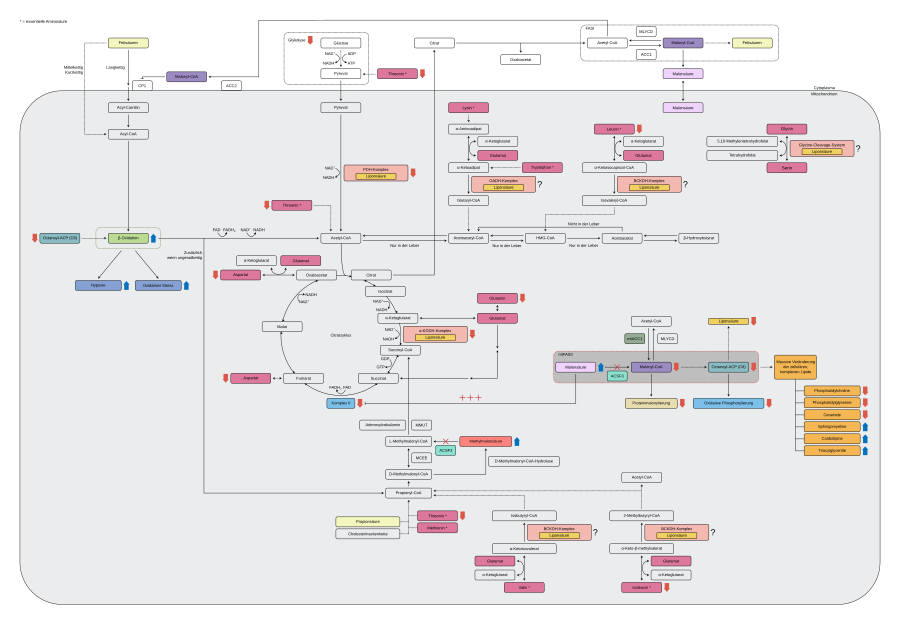
Defekt der mitochondrialen Fettsäuresynthese (mtFASII)
ACSF3 katalysiert in seiner Funktion als Malonyl-CoA-Synthetase die Umwandlung von Malonsäure in Malonyl-CoA, welches den ersten Schritt der mitochondrialen Fettsäuresynthese (mtFASII) darstellt.[9][3] Die mtFASII – nicht zu verwechseln mit der bekannteren Fettsäuresynthese (FASI) im Cytoplasma – spielt eine wichtige Rolle bei der Regulierung des Energiestoffwechsels und bei lipidvermittelten Signalprozessen.[11][3]
Der Mangel an ACSF3 bei CMAMMA führt zu einer Anhäufung von Malonsäure und einem mitochondrialen Mangel an Malonyl-CoA.[12] Während Malonsäure den Komplex II kompetitiv hemmt und zytotoxisch wirkt, führt der Mangel am Substrat Malonyl-CoA wiederum zu einer verminderten Malonylierung mitochondrialer Proteine, was die Aktivität von Stoffwechselenzymen beeinflusst und den Zellstoffwechsel verändert.[11] Der Malonyl-CoA-Bedarf kann jedoch über das Enzym mtACC1, einer mitochondriale Isoform von ACC1, zum Teil gedeckt werden, was den breiten klinischen Phänotyp von CMAMMA erklärt.[13] Die Defizite an Zwischenprodukten lassen sich bis zum Hauptprodukt von mtFASII, Octanoyl-ACP, weiterführen, das als Ausgangssubstrat für die Biosynthese von Liponsäure, für die Assemblierung der Komplexe der oxidativen Phosphorylierung und als endogenes Substrat für die β-Oxidation benötigt wird.[3] Wichtige mitochondriale Multienzymkomplexe wie die des Energiestoffwechsels, der Pyruvat-Dehydrogenase-Komplex (PDHC), der α-Ketoglutarat-Dehydrogenase-Komplex (α-KGDHC) und des Aminosäurenstoffwechsels, der verzweigtkettige Alpha-Ketosäure-Dehydrogenase-Komplex (BCKDHC), der Oxoadipat-Dehydrogenase-Komplex (OADHC) und das Glycin-Cleavage-System (GCS), sind für ihre Funktionalität auf Liponsäure als kovalenten Cofaktor angewiesen.[14][15] Infolgedessen führt die verringerte Lipoylierung des Pyruvat-Dehydrogenase-Komplexes und des α-Ketoglutarat-Dehydrogenase-Komplexes zu einem verringerten glykolytischen Fluss, gemessen in Glykolyse und glykolytischer Kapazität.[3] Um wahrscheinlich den Energiebedarf der Zelle zu kompensieren, konnte eine Hochregulierung der Fettsäure-β-Oxidation und eine verringerte Konzentration von Aminosäuren, die anaplerotisch in den Citratzyklus einfließen, wie Aspartat, Glutamin, Isoleucin, Threonin und Leucin festgestellt werden.[3] In der Zusammenfassung führt die reduzierte mitochondriale Atmung und der glykolytische Fluss zu einer beeinträchtigten mitochondrialen Flexibilität mit einer großen Abhängigkeit von der Fettsäure-β-Oxidation und einem erhöhten Verbrauch von anaplerotischen Aminosäuren.[3][13]
Jedoch sind Nervenzellen trotz ihres hohen Energiebedarfs nicht in der Lage, Fettsäuren effizient zur Energiegewinnung zu nutzen, mit Ausnahme von Gliazellen und spezialisierten Neuronen im Hypothalamus.[13] Dennoch besteht eine enge metabolische Interaktion zwischen Gliazellen in Form von Astrozyten und Neuronen zur Aufrechterhaltung der zellulären Funktionalität.[13] Es wird daher spekuliert, dass CMAMMA auch zu einer Hochregulierung der β-Oxidation in den Gehirnzellen führt, mit der Folge eines erhöhten Risikos für Hypoxie und oxidativen Stress, was langfristig zu neurologischen Symptomen beitragen kann.[13]
Zusätzlich wird spekuliert, dass die verringerte neuronale ATP-Erzeugung zu einer verringerten Wiederaufnahme von Glutamat aus dem synaptischen Spalt führt, wenn nicht sogar zu einer kompletten Umkehrung des Glutamattransportes, was in einer Erhöhung der intrazellulären Ca+2-Konzentration und einer Depolarization der spannungsabhängigen Na+- und Ca+-Kanäle resultiert.[16] Die Folgen durch die unspezifische Stimulierung des erhöhten, extrazellulären Glutamates an den ionotropischen Glutamatrezeptoren AMPA und NMDA sind neurolale Schäden. In In-vitro-Modellen konnte dieser beschriebene Pathomechanismus durch den Einsatz von Kreatin, den NMDA-Rezeptorantagonisten MK-801, Ifenprodil und dem AMPA-Rezeptorantagonist CNQX beeinflusst werden.[16]
Des Weiteren kommt es zu massiven Veränderungen der zellulären komplexen Lipide, so sind die Werte bioaktiver Lipide wie Sphingomyeline und Cardiolipine, als auch Triacylglyceride, die zusätzlich mit einer veränderten Fettsäurekettenlänge und dem Vorhandensein ungeradkettiger Spezies einhergehen.[3] Im Gegensatz dazu die Werte von Phosphatidylcholine, Phosphatidylglycerine und Ceramide reduziert, letztere proportional zum Anstieg der Sphingomyeline.[3] Darüber hinaus ist der Einbau von Malonat in Lipide deutlich veringert, was darauf hindeutet, dass ACSF3 für den Malonat-Stoffwechsel erforderlich ist.[12]
Defekt des Methylmalonsäureabbaus (Methylmalonazidurie)
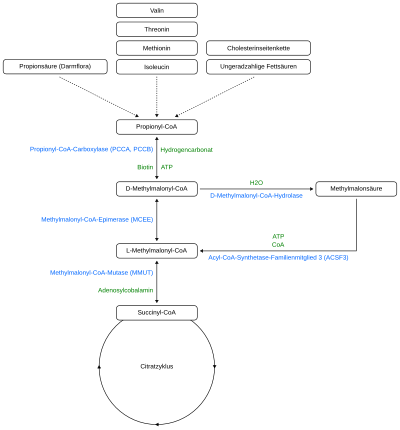
ACSF3 katalysiert in seiner Funktion als Methylmalonyl-CoA-Synthetase die Umwandlung von Methylmalonsäure in Methylmalonyl-CoA, damit diese über den Zitronensäurezyklus abgebaut werden kann.
Der Mangel an ACSF3 bei CMAMMA führt daher zu einem verminderten Abbau und folglich zu einer erhöhten Anhäufung von Methylmalonsäure in Körperflüssigkeiten und Geweben, was auch als Methylmalonazidurie bekannt ist. Methylmalonyl-CoA wird aus den essentiellen Aminosäuren Valin, Threonin, Methionin und Isoleucin, aus ungeradzahligen Fettsäuren, aus Propionsäure und aus der Cholesterinseitenkette gebildet und kann von der D-Methylmalonyl-CoA-Hydrolase in Methylmalonsäure umgewandelt werden, noch bevor es die Succinyl-CoA-Stelle des Citratzykluses erreicht.
Die bakterielle Fermentation im Darm stellt über die Vorstufe Propionsäure, eine quantitativ signifikante Quelle für Methylmalonsäure dar.[17] Daneben wird Propionsäure aber auch über die Ernährung aufgenommen, da es in bestimmten Lebensmitteln natürlich enthalten ist oder als Konservierungsstoff durch die Lebensmittelindustrie beigefügt wird, vor allem bei Backwaren[18] und Molkereiprodukten.[19] Darüber hinaus entsteht Methylmalonat bei dem Thymin-Katabolismus.[17]
Jedoch sind auch intrazelluläre Esterasen fähig die Methylgruppe (-CH3) der Methylmalonsäure abzuspalten und das Ausgangsmolekül Malonsäure zu generieren.[20]
In vitro konnte eine Verbindung zwischen freier Methylmalonsäure und Malonsäure zu Neurotoxizität hergestellt werden.[16][20]
Diagnostik
Aufgrund der vielfältigen klinischen Symptome und der Tatsache, dass die CMAMMA weitgehend unentdeckt durch Neugeborenen-Screening-Programme hindurchgeht, gilt die CMAMMA als eine nur unzureichend erkannte Erkrankung.[1][2]
Neugeborenen-Screening-Programme
Da es bei CMAMMA nicht zu einer Anhäufung von Methylmalonyl-CoA, Malonyl-CoA oder Propionyl-CoA kommt und sich auch keine Anomalien im Acylcarnitinprofil zeigen, wird die CMAMMA durch die üblichen blutbasierten Neugeborenenscreeningprogramme nicht erkannt.[4][6][2]
Ein Sonderfall stellt die Provinz Quebec dar, in der im Rahmen des Quebec Neonatal Blood and Urine Screening Program, zusätzlich zum Bluttest, am 21. Tag nach der Geburt noch der Urin untersucht wird, obwohl es wahrscheinlich ist, dass hierbei nicht jeder mit CMAMMA entdeckt wird.[21][6]
Routine- und biochemische Laboruntersuchungen
Für die Bestimmung des Verhältnisses von Malonsäure zu Methylmalonsäure wird die Verwendung von Blutplasma anstelle von Urin empfohlen. Denn selbst bei einer Erhöhung der labortechnischen Sensitivität für Malonsäure (MA) können die Konzentrationen in Urinproben so gering sein, dass diese trotzdem leicht übersehen werden. Wird dagegen nur Methylmalonsäure (MAA) im Urin als alleinige Matrix verwendet, besteht die Möglichkeit, dass eine CMAMMA als klassische Methylmalonazidurie fehldiagnostiziert wird. Auch die Berechnung des MA/MAA-Verhältnisses im Urin ist nicht sinnvoll, da aufgrund von Überschneidungen mit gesunden Personen nicht eindeutig von CMAMMA-Betroffenen unterschieden werden kann. Hingegen kann die Berechnung des MA/MMA-Verhältnisses im Plasma eine CMAMMA eindeutig von einer klassischen Methylmalonazidurie unterscheiden. Dies gilt sowohl für Vitamin-B12-Responder als auch für Non-Responder der Methylmalonazidurie.[1]
Bei CMAMMA übersteigen hierbei die Methylamlonsäurewerte die der Malonsäure. Im Gegensatz dazu ist bei der Malonazidurie das MMA/MA-Verhältnis kleiner als 1.[8][2]
Molekulargenetische Untersuchung
Die endgültige Diagnose wird durch molekulargenetische Untersuchungen bestätigt, wenn biallelische pathogene Varianten im ACSF3-Gen gefunden werden. Hierzu gibt spezifische Multigen-Panels für Methylmalonazidurien, aber die getesteten Gene können von Labor zu Labor variieren und können vom Kliniker an den individuellen Phänotyp angepasst werden.[22][23]
Im Rahmen von Fertilitätsbehandlungen können durch ein erweitertes Trägerscreening (ECS) auch Träger von Mutationen im Gen ACSF3 identifiziert werden.[24]
Behandlung
Ernährung
Ein Ansatz zur Verringerung der sich ansammelnden Malonsäure- und Methylmalonsäuremenge ist die Ernährung. Hier hat sich eine kohlenhydratreiche und eiweißarme Ernährung als am besten erwiesen. Veränderungen der Malonsäure- und Methylmalonsäureausscheidung lassen sich bereits 24 bis 36 Stunden nach einer Ernährungsumstellung feststellen.[8]
Bakterienreduzierende Maßnahmen
Eine weitere quantitativ bedeutsame Quelle für Malonsäure und Methylmalonsäure ist neben der Aufnahme von Eiweiß aus der Nahrung, die bakterielle Fermentation.[17] Davon abgeleitet ergeben sich Behandlungsmaßnahmen wie die Gabe von Antibiotika und Abführmitteln.
Vitamin B12
Da einige Formen der Methylmalonazidurie auf Vitamin B12 ansprechen, wurden bei CMAMMA, Behandlungsversuche mit Vitamin B12 unternommen, auch in Form von Hydroxocobalamin-Injektionen, die jedoch zu keinen klinischen oder biochemischen Effekten führten.[1][2]
L-Carnitin
In einer Studie wird auch die Behandlung mit L-Carnitin bei Patienten mit CMAMMA erwähnt, allerdings nur retrospektiv und ohne Nennung von Ergebnissen.[2]
Messenger RNA
Präklinische Proof-of-Concept-Studien an Tiermodellen haben gezeigt, dass sich messenger RNA (mRNA)-Therapien auch für den Einsatz bei seltenen Stoffwechselerkrankungen eignen.[25] In diesem Zusammenhang ist die seit 2019 laufende Phase-1/2-Studie (mRNA-3704 & mRNA-3705) des Biotechnologieunternehmens Moderna zur Behandlung der isolierten Methylmalonazidurie erwähnenswert.[26][27]
Forschung
1984 wurde die CMAMMA aufgrund eines Malonyl-CoA-Decarboxylase-Mangels erstmals in einer wissenschaftlichen Studie beschrieben.[8][28] Weitere Studien zu dieser Form von CMAMMA folgten bis 1994, als eine andere Form der CMAMMA mit normaler Malonyl-CoA-Decarboxylase-Aktivität entdeckt wurde.[8][29] Im Jahr 2011 identifizierte die genetische Forschung durch Exom-Sequenzierung das Gen ACSF3 als Ursache für CMAMMA mit normaler Malonyl-CoA-Decarboxylase.[4][6] Mit einer 2016 veröffentlichten Studie wurde die Berechnung des MA/MAA-Verhältnisses im Plasma als eine neue Möglichkeit zur schnellen, metabolischen Diagnose der CMAMMA vorgestellt.[1]
Das Quebec Neonatal Blood and Urine Screening Program macht die Provinz Quebec für die CMAMMA-Forschung interessant, da es die einzige Patientenkohorte der Welt ohne Selektionsverzerrungen darstellt.[2] Zwischen 1975 und 2010 wurden auf diese Weise schätzungsweise 2 695 000 Neugeborene untersucht, hierbei wurden 3 Fälle von CMAMMA festgestellt.[2] Da die Entdeckungsrate jedoch niedriger ist als die aufgrund der heterozygoten Häufigkeit vorhergesagte Rate ist, ist es wahrscheinlich, dass nicht alle Neugeborenen mit diesem biochemischen Phänotyp durch das Screening-Programm entdeckt wurden.[6] In einer Studie aus dem Jahr 2019 wurden in der Provinz Quebec schon 25 Patienten mit CMAMMA identifiziert. Bis auf einen wurden alle durch das Provincial Neonatal Urine Screening Program entdeckt, 20 davon direkt und 4 nach der Diagnose eines älteren Geschwisterteils.[2]
Phänotypische Serie
Auch bei den folgenden Krankheiten ist der Gehalt an Malonsäure und Methylmalonsäure biochemisch erhöht:
Siehe auch
Anmerkungen
In der wissenschaftlichen Literatur hat sich der Begriff der kombinierten Malon- und Methylmalonazidurie mit dem Suffix -urie (von griechisch ouron, Urin) im Gegensatz zum anderen Begriff der kombinierten Malon- und Methylmalonazidämie mit dem Suffix -ämie (von griechisch aima, Blut) durchgesetzt. Im Zusammenhang mit der CMAMMA wird jedoch keine klare Unterscheidung getroffen, da Malonsäure und Methylmalonsäure sowohl im Blut als auch im Urin erhöht sind.
Bei der Malonazidurie sind auch Malonsäure und Methylmalonsäure erhöht, weshalb sie früher auch als kombinierte Malon- und Methylmalonazidurie (CMAMMA) bezeichnet wurde. Obwohl der ACSF3-Mangel erst später entdeckt wurde, hat sich der Begriff der kombinierten Malon- und Methylmalonazidurie in medizinischen Datenbanken für den ACSF3-Mangel durchgesetzt.[30][31]
Weblinks
- Kombinierte Malon- und Methylmalonazidurie auf Orphanet
- Combined malonic and methylmalonic aciduria auf OMIM
- Combined malonic and methylmalonic aciduria auf PatientsLikeMe
Einzelnachweise
- ↑ a b c d e f Monique G. M. de Sain-van der Velden, Maria van der Ham, Judith J. Jans, Gepke Visser, Hubertus C. M. T. Prinsen: A New Approach for Fast Metabolic Diagnostics in CMAMMA. In: JIMD Reports. Band 30. Springer Berlin Heidelberg, Berlin, Heidelberg 2016, ISBN 978-3-662-53680-3, S. 15–22, doi:10.1007/8904_2016_531, PMID 26915364, PMC 5110436 (freier Volltext).
- ↑ a b c d e f g h i j k l m Alina Levtova, Paula J. Waters, Daniela Buhas, Sébastien Lévesque, Christiane Auray‐Blais: Combined malonic and methylmalonic aciduria due to ACSF3 mutations: Benign clinical course in an unselected cohort. In: Journal of Inherited Metabolic Disease. Band 42, Nr. 1, Januar 2019, ISSN 0141-8955, S. 107–116, doi:10.1002/jimd.12032.
- ↑ a b c d e f g h i j k l Zeinab Wehbe, Sidney Behringer, Khaled Alatibi, David Watkins, David Rosenblatt: The emerging role of the mitochondrial fatty-acid synthase (mtFASII) in the regulation of energy metabolism. In: Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. Band 1864, Nr. 11, November 2019, S. 1629–1643, doi:10.1016/j.bbalip.2019.07.012 (elsevier.com).
- ↑ a b c d e f g h i j k l m n NIH Intramural Sequencing Center Group, Jennifer L Sloan, Jennifer J Johnston, Irini Manoli, Randy J Chandler: Exome sequencing identifies ACSF3 as a cause of combined malonic and methylmalonic aciduria. In: Nature Genetics. Band 43, Nr. 9, September 2011, ISSN 1061-4036, S. 883–886, doi:10.1038/ng.908, PMID 21841779, PMC 3163731 (freier Volltext).
- ↑ Lisa C. Sniderman, Marie Lambert, Robert Giguère, Christiane Auray-Blais, Bernard Lemieux: Outcome of individuals with low-moderate methylmalonic aciduria detected through a neonatal screening program. In: The Journal of Pediatrics. Band 134, Nr. 6, Juni 1999, S. 675–680, doi:10.1016/S0022-3476(99)70280-5 (elsevier.com).
- ↑ a b c d e f g h i j k l m A. Alfares, L. D. Nunez, K. Al-Thihli, J. Mitchell, S. Melancon: Combined malonic and methylmalonic aciduria: exome sequencing reveals mutations in the ACSF3 gene in patients with a non-classic phenotype. In: Journal of Medical Genetics. Band 48, Nr. 9, 1. September 2011, ISSN 0022-2593, S. 602–605, doi:10.1136/jmedgenet-2011-100230.
- ↑ a b c d Ping Wang, Jianbo Shu, Chunyu Gu, Xiaoli Yu, Jie Zheng: Combined Malonic and Methylmalonic Aciduria Due to ACSF3 Variants Results in Benign Clinical Course in Three Chinese Patients. In: Frontiers in Pediatrics. Band 9, 25. November 2021, ISSN 2296-2360, S. 751895, doi:10.3389/fped.2021.751895, PMID 34900860, PMC 8658908 (freier Volltext).
- ↑ a b c d e f g h i j A. R. Gregg, A. W. Warman, D. R. Thorburn, W. E. OʼBrien: Combined malonic and methylmalonic aciduria with normal malonyl-coenzyme A decarboxylase activity: A case supporting multiple aetiologies. In: Journal of Inherited Metabolic Disease. Band 21, Nr. 4, Juni 1998, S. 382–390, doi:10.1023/A:1005302607897.
- ↑ a b Andrzej Witkowski, Jennifer Thweatt, Stuart Smith: Mammalian ACSF3 Protein Is a Malonyl-CoA Synthetase That Supplies the Chain Extender Units for Mitochondrial Fatty Acid Synthesis. In: Journal of Biological Chemistry. Band 286, Nr. 39, September 2011, S. 33729–33736, doi:10.1074/jbc.M111.291591, PMID 21846720, PMC 3190830 (freier Volltext) – (elsevier.com).
- ↑ Joon Kee Lee, Arum Oh: Combined Malonic and Methylmalonic Aciduria Diagnosed by Recurrent and Severe Infections Mimicking a Primary Immunodeficiency Disease: A Case Report. In: Journal of Korean Medical Science. Band 38, Nr. 45, 2023, ISSN 1011-8934, doi:10.3346/jkms.2023.38.e387, PMID 37987109, PMC 10659923 (freier Volltext).
- ↑ a b Caitlyn E. Bowman, Michael J. Wolfgang: Role of the malonyl-CoA synthetase ACSF3 in mitochondrial metabolism. In: Advances in Biological Regulation. Band 71, Januar 2019, S. 34–40, doi:10.1016/j.jbior.2018.09.002, PMID 30201289, PMC 6347522 (freier Volltext) – (elsevier.com).
- ↑ a b Caitlyn E. Bowman, Susana Rodriguez, Ebru S. Selen Alpergin, Michelle G. Acoba, Liang Zhao, Thomas Hartung, Steven M. Claypool, Paul A. Watkins, Michael J. Wolfgang: The Mammalian Malonyl-CoA Synthetase ACSF3 Is Required for Mitochondrial Protein Malonylation and Metabolic Efficiency. In: Cell Chemical Biology. Band 24, Nr. 6, Juni 2017, S. 673–684.e4, doi:10.1016/j.chembiol.2017.04.009, PMID 28479296, PMC 5482780 (freier Volltext).
- ↑ a b c d e Sara Tucci: Brain metabolism and neurological symptoms in combined malonic and methylmalonic aciduria. In: Orphanet Journal of Rare Diseases. Band 15, Nr. 1, Dezember 2020, ISSN 1750-1172, S. 27, doi:10.1186/s13023-020-1299-7, PMID 31969167, PMC 6977288 (freier Volltext).
- ↑ Johannes A. Mayr, René G. Feichtinger, Frederic Tort, Antonia Ribes, Wolfgang Sperl: Lipoic acid biosynthesis defects. In: Journal of Inherited Metabolic Disease. Band 37, Nr. 4, Juli 2014, ISSN 0141-8955, S. 553–563, doi:10.1007/s10545-014-9705-8.
- ↑ Geoffray Monteuuis, Fumi Suomi, Juha M. Kerätär, Ali J. Masud, Alexander J. Kastaniotis: A conserved mammalian mitochondrial isoform of acetyl-CoA carboxylase ACC1 provides the malonyl-CoA essential for mitochondrial biogenesis in tandem with ACSF3. In: Biochemical Journal. Band 474, Nr. 22, 15. November 2017, ISSN 0264-6021, S. 3783–3797, doi:10.1042/BCJ20170416 (portlandpress.com).
- ↑ a b c S. Kölker, B. Ahlmeyer, J. Krieglstein, G. F. Hoffmann: Methylmalonic acid induces excitotoxic neuronal damage in vitro. In: Journal of Inherited Metabolic Disease. Band 23, Nr. 4, Juni 2000, S. 355–358, doi:10.1023/A:1005631230455.
- ↑ a b c G.N. Thompson, J.H. Walter, J.-L. Bresson, G.C. Ford, S.L. Lyonnet: Sources of propionate in inborn errors of propionate metabolism. In: Metabolism. Band 39, Nr. 11, November 1990, S. 1133–1137, doi:10.1016/0026-0495(90)90084-P (elsevier.com).
- ↑ Marwa Scharinger, Marcel Kuntz, Andreas Scharinger, Jan Teipel, Thomas Kuballa: Rapid Approach to Determine Propionic and Sorbic Acid Contents in Bread and Bakery Products Using 1H NMR Spectroscopy. In: Foods. Band 10, Nr. 3, 3. März 2021, ISSN 2304-8158, S. 526, doi:10.3390/foods10030526, PMID 33802459, PMC 7998730 (freier Volltext) – (mdpi.com).
- ↑ T Yamamura, Y Okamoto, G Okada, Y Takaishi, M Takamura: Association of thalamic hyperactivity with treatment-resistant depression and poor response in early treatment for major depression: a resting-state fMRI study using fractional amplitude of low-frequency fluctuations. In: Translational Psychiatry. Band 6, Nr. 3, März 2016, ISSN 2158-3188, S. e754–e754, doi:10.1038/tp.2016.18, PMID 26954981, PMC 4872444 (freier Volltext).
- ↑ a b B.A McLaughlin, D Nelson, I.A Silver, M Erecinska, M.-F Chesselet: Methylmalonate toxicity in primary neuronal cultures. In: Neuroscience. Band 86, Nr. 1, Mai 1998, S. 279–290, doi:10.1016/S0306-4522(97)00594-0 (elsevier.com).
- ↑ Blood and Urine Screening in Newborns. Abgerufen am 8. Juli 2022 (englisch).
- ↑ a b c Manoli I, Sloan JL, Venditti CP. Isolated Methylmalonic Acidemia. 2005 Aug 16 [Updated 2022 Sep 8]. In: Adam MP, Feldman J, Mirzaa GM, et al., editors. GeneReviews® [Internet]. Seattle (WA): University of Washington, Seattle; 1993–2024. Available from: https://www.ncbi.nlm.nih.gov/books/NBK1231/
- ↑ Targeted Genes and Methodology Details for Methylmalonic Aciduria Gene Panel. Mayo Clinic Laboratories, abgerufen am 16. Mai 2024 (englisch).
- ↑ Marie Cosette Gabriel, Stephanie M. Rice, Jennifer L. Sloan, Matthew H. Mossayebi, Charles P. Venditti: Considerations of expanded carrier screening: Lessons learned from combined malonic and methylmalonic aciduria. In: Molecular Genetics & Genomic Medicine. Band 9, Nr. 4, April 2021, ISSN 2324-9269, doi:10.1002/mgg3.1621, PMID 33625768, PMC 8123733 (freier Volltext).
- ↑ Paolo G.V. Martini, Lin T. Guey: A New Era for Rare Genetic Diseases: Messenger RNA Therapy. In: Human Gene Therapy. Band 30, Nr. 10, 1. Oktober 2019, ISSN 1043-0342, S. 1180–1189, doi:10.1089/hum.2019.090.
- ↑ A Clinical Trial of a Treatment for Patients with Methylmalonic Acidemia (MMA). Abgerufen am 8. Juli 2022.
- ↑ A Clinical Trial of a Treatment for Patients with Methylmalonic Acidemia (MMA). Abgerufen am 8. Juli 2022.
- ↑ G. K. Brown, R. D. Scholem, A. Bankier, D. M. Danks: Malonyl coenzyme a decarboxylase deficiency. In: Journal of Inherited Metabolic Disease. Band 7, Nr. 1, März 1984, ISSN 0141-8955, S. 21–26, doi:10.1007/BF01805615.
- ↑ P.T. Ozand, W.L. Nyhan, A. Al Aqeel, J. Christodoulou: Malonic aciduria. In: Brain and Development. Band 16, November 1994, S. 7–11, doi:10.1016/0387-7604(94)90091-4 (elsevier.com).
- ↑ COMBINED MALONIC AND METHYLMALONIC ACIDURIA; CMAMMA. In: OMIM. Abgerufen am 24. April 2024.
- ↑ Combined malonic and methylmalonic acidemia. In: National Library of Medicine. Abgerufen am 24. April 2024.




