Klonierungsvektor
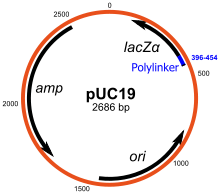
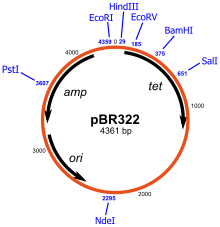
Ein Klonierungsvektor ist ein Vektor, der im Gegensatz zum Expressionsvektor nur zur Klonierung verwendet wird.
Eigenschaften
Klonierungsvektoren besitzen einen Replikationsursprung für die Replikation der DNA, eine Sequenz zur Selektion des Expressionsvektors (z. B. eine Antibiotikumresistenz oder eine Auxotrophie) und einen Polylinker, in die eine zu klonierende DNA-Sequenz (das Insert) eingefügt wird.[1] Meistens werden Klonierungsvektoren in Form eines Plasmids verwendet, aber auch Bacterial Artificial Chromosomes und λ-Vektoren und M13-Vektoren.[2] Als Antibiotikaresistenzen werden Resistenzgene gegen Ampicillin, Chloramphenicol, Kanamycin und Tetracycline verwendet.
Klonierungsvektoren werden verwendet, um die DNA des Inserts in Bakterien zu vermehren oder um die Restriktionsschnittstellen des Inserts über den Polylinker zu ändern. Dabei war die Änderung der Restriktionsstellen durch Klonierungsvektoren die erste verfügbare Methode zur Änderung der Restriktionsstellen, während zunehmend andere Methoden zur Änderung der Restriktionsschnittstelle wie die PrimerExtension-PCR oder restriktionsfreie Methoden der Klonierung (z. B. TOPO-Klonierung oder Gateway-Klonierung) verwendet werden.
Literatur
- M. P. Mayer: A new set of useful cloning and expression vectors derived from pBlueScript. In: Gene. Band 163, Nummer 1, September 1995, S. 41–46, PMID 7557476.
- B. Rohweder, F. Semmelmann, C. Endres, R. Sterner: Standardized cloning vectors for protein production and generation of large gene libraries in Escherichia coli. In: BioTechniques. Band 64, Nummer 1, Januar 2018, S. 24–26, doi:10.2144/000114628, PMID 29384074.
Einzelnachweise
- ↑ Mary Campbell: Biochemistry. Cengage Learning, 2007, ISBN 978-0-495-39041-1, S. 378.
- ↑ Nicola Casali: E. Coli Plasmid Vectors. Springer Science & Business Media, 2003, ISBN 978-1-592-59409-2, S. 20.




