„Desoxyribonukleinsäure“ – Versionsunterschied
| [ungesichtete Version] | [ungesichtete Version] |
→Aufbau der DNA: angefangen, textlich durchzugehen, aber nur bis Base gekommen. Vieles ist noch doppelt. |
K Bot: Ergänze: nov:DNA |
||
| Zeile 31: | Zeile 31: | ||
[[Bild:DNA As Structure Formula (German).PNG|thumb|370px|Schematische Darstellung eines DNA-Ausschnittes mit zwei Basenpaaren. Bindungslängen und -winkel sind nicht maßstabsgetreu.]] |
[[Bild:DNA As Structure Formula (German).PNG|thumb|370px|Schematische Darstellung eines DNA-Ausschnittes mit zwei Basenpaaren. Bindungslängen und -winkel sind nicht maßstabsgetreu.]] |
||
[[Image:PhosphodiesterBondDiagram.png|thumb|Zwei Phosphodiester Bindungen (blaue Kreise), die insgesamt drei Nukleotide verbinden.]] |
[[Image:PhosphodiesterBondDiagram.png|thumb|Zwei Phosphodiester Bindungen (blaue Kreise), die insgesamt drei Nukleotide verbinden.]] |
||
Die Desoxyribonukleinsäure ist ein langes Kettenmolekül ([[Polymer]]) aus vielen Einzelbausteinen, die man [[Nukleotid|Desoxyribonukleotide]] |
Die Desoxyribonukleinsäure ist ein langes Kettenmolekül ([[Polymer]]) aus vielen Einzelbausteinen, die man [[Nukleotid|Desoxyribonukleotide]] nennt. Jedes Nukleotid ist eine Verbindung aus einem [[Phosphorsäure]]-Molekül, dem [[Zucker]] [[Desoxyribose]] sowie einer [[Heterocyclen|heterozyklischen]] [[Nukleinbasen|Nukleobase]]: [[Adenin]] ('''A'''), [[Thymin]] ('''T'''), [[Guanin]] ('''G''') oder [[Cytosin]] ('''C'''). Die Desoxyribose- und Phosphorsäure-Untereinheiten sind bei jedem Nukleotid gleich und werden auch als "Rückgrat" des Moleküls bezeichnet; die vier verschiedenen Nukleotide unterscheiden sich also nur durch ihre Base und entsprechend werden die Abkürzungen A, T, G und C auch für die entsprechenden Nukleotide verwendet. |
||
Die fünf Kohlenstoffatome einer |
Die fünf Kohlenstoffatome einer Desoxyribose sind von 1' (sprich ''Eins Strich'') bis 5' nummeriert. Am 1'-Ende dieses Zuckers sitzt die Nukleobase und am 5'-Ende hängt der [[Phosphorsäure]]- oder Phosphatrest. Eigentlich handelt es sich bei der Desoxyribose um die 2-Desoxyribose, da die [[OH-Gruppe]] (Alkohol) an der Position 2' fehlt (bzw. durch einen Wasserstoff ersetzt wurde). An der Position 3' jedoch befindet sich immer noch eine OH-Gruppe, welche die Desoxirobose über eine Phosphatgruppe zum 5' Kohlenstoffatom des jeweils nächsten Zuckers verknüpft, eine sogenannte Phosphodiester Bindung (siehe Abbildung). Somit besitzt jedes DNA-Molekül zwei verschiedene Enden: Ein 5' Ende und ein 3' Ende. Neue Bausteine werden immer an die OH-Gruppe am 3' Ende angefügt (die Kette kann also nur an einem Ende verlängert werden, dies hat Implikationen in der DNA-Replikation), indem ein [[Nukleosidtriphosphat]]e (mit drei Phosphatresten) als neuer Baustein angeliefert wird und zwei Phosphate in Form von [[Pyrophosphat]] entfernt werden und der verbleibende Phosphatrest des jeweils neu hinzukommenden Nukleotids mit der OH-Gruppe am 3'-Ende des letzten im Strang vorhandenen Nucleotids unter Wasserabspaltung reagiert. Auf diese Weise können zahlreiche weitere Nukleotide an das 3'-Ende eines DNA-Einzelstrangs angehängt werden. |
||
Jede Desoxyribose hat an ihrer 1' Position eine |
Jede Desoxyribose hat an ihrer 1' Position eine Base gebunden (A, G, C oder G), deren Abfolge schlussendlich die genetische Information kodieren. Während sich die Desoxyribosen verbunden durch Phosphat wie die "Stränge" einer Strickleiter winden, stellen die Basen dabei die "Sprossen" der Leiter dar. Dabei werden immer zwei Basen gepaart, jeweils ein [[Purin]] (Adenin bzw. Guanin) mit einem [[Pyrimidin]] (Thymin bzw. Cytosin ). Zwischen Adenin und Thymin bilden sich dabei zwei [[Wasserstoffbrücke]]n aus; Cytosin und Guanin sind über drei Wasserstoffbrücken miteinander verknüpft. Es bilden sich also in den "Sprossen" keine Atombindungen aus sondern lediglich schwache Wasserstoffbrücken, was es ermöglicht, die beiden Stränge auch wieder zu trennen. Dadurch, dass immer ein Purin mit einem Pyrimidin kombiniert wird, bleibt der Abstand zwischen den Strängen (also die Länge der "Sprossen") gleich und eine regelmässige Struktur kann entstehen. |
||
Normalerweise ist DNA rechtshändig gedreht mit den oben genannten Strukturmerkmalen. Neben dieser, auch „B-DNA“ genannten [[Konformation]], existieren auch eine „A-Form“ sowie eine 1979 von [[Alexander Rich]] und seinen Kollegen am [[Massachusetts Institute of Technology|MIT]] erstmals auch untersuchte, linkshändige, so genannte „[[Z-DNA]]“. Diese tritt besonders in G-C-reichen Abschnitten auf. Erst 2005 wurde über eine Kristallstruktur berichtet, welche Z-DNA direkt in einer Verbindung mit B-DNA zeigt und so Hinweise auf eine biologische Aktivität von Z-DNA liefert. |
Normalerweise ist DNA rechtshändig gedreht mit den oben genannten Strukturmerkmalen. Neben dieser, auch „B-DNA“ genannten [[Konformation]], existieren auch eine „A-Form“ sowie eine 1979 von [[Alexander Rich]] und seinen Kollegen am [[Massachusetts Institute of Technology|MIT]] erstmals auch untersuchte, linkshändige, so genannte „[[Z-DNA]]“. Diese tritt besonders in G-C-reichen Abschnitten auf. Erst 2005 wurde über eine Kristallstruktur berichtet, welche Z-DNA direkt in einer Verbindung mit B-DNA zeigt und so Hinweise auf eine biologische Aktivität von Z-DNA liefert. |
||
<ref name=Ha2005>{{cite journal |author=Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK |title=Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases |journal=Nature |volume=437 |pages=1183-1186 |year=2005}} PMID 16237447 </ref> |
<ref name=Ha2005>{{cite journal |author=Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK |title=Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases |journal=Nature |volume=437 |pages=1183-1186 |year=2005}} PMID 16237447 </ref> |
||
Die Tabelle unten und die daneben stehende Abbildung zeigen die Unterschiede der drei Formen im direkten Vergleich. |
|||
{| align=right |
{| align=right |
||
| Zeile 45: | Zeile 46: | ||
||[[Bild:DNA.jpg|thumb|none|Ausschnitt aus einem [[Kalottenmodell]] eines DNA-Moleküls. Während andere Modelle gut geeignet sind, die Beziehung der einzelnen Atome zueinander darzustellen, zeigt ein Kalottenmodell die Belegung des Raumvolumens. Es wird daher der falsche Eindruck vermieden, dass zwischen den einzelnen Atomen noch viel Raum sei.]] |
||[[Bild:DNA.jpg|thumb|none|Ausschnitt aus einem [[Kalottenmodell]] eines DNA-Moleküls. Während andere Modelle gut geeignet sind, die Beziehung der einzelnen Atome zueinander darzustellen, zeigt ein Kalottenmodell die Belegung des Raumvolumens. Es wird daher der falsche Eindruck vermieden, dass zwischen den einzelnen Atomen noch viel Raum sei.]] |
||
|} |
|} |
||
Es existieren drei Strukturmodelle der DNA (die A, B und Z-DNA) wobei die biologisch wichtigste die "B-Form" ist, wie sie auch von Watson und Crick vorgeschlagen wurde. Hier wird nur auf die B-Form eingegangen, die Tabelle weiter unten zeigt die Unterschiede der drei Formen im direkten Vergleich. |
|||
Die DNA besteht aus zwei langen Molekülketten, die schraubenförmig um eine gemeinsame, gedachte Achse gewunden sind ([[Doppelhelix]]-Struktur). Die Helix besteht aus einem Rückgrat aus alternierenden [[Phosphate|Phosphatresten]] und [[Kohlenhydrate|Zuckerresten]] (2-Desoxribosen), an welche jeweils die zum Zentrum der Helix gerichteten Basen gebunden sind. |
Die DNA besteht aus zwei langen Molekülketten, die schraubenförmig um eine gemeinsame, gedachte Achse gewunden sind ([[Doppelhelix]]-Struktur). Die Helix besteht aus einem Rückgrat aus alternierenden [[Phosphate|Phosphatresten]] und [[Kohlenhydrate|Zuckerresten]] (2-Desoxribosen), an welche jeweils die zum Zentrum der Helix gerichteten Basen gebunden sind. |
||
Der Drehsinn der beiden Ketten (DNA-Einzelstränge) ist in der B-DNA jeweils rechts und die beiden Ketten sind antiparallel, sie besitzen also jeweils eine ''komplementäre'' Basensequenz. Die ganze Helix hat einen Durchmesser von ungefähr 2 nm und windet sich mit jedem Zuckermoleküle um 0.34 nm nach oben. |
Der Drehsinn der beiden Ketten (DNA-Einzelstränge) ist in der B-DNA jeweils rechts und die beiden Ketten sind antiparallel, sie besitzen also jeweils eine ''komplementäre'' Basensequenz. Die ganze Helix hat einen Durchmesser von ungefähr 2 nm und windet sich mit jedem Zuckermoleküle um 0.34 nm nach oben. |
||
| Zeile 257: | Zeile 261: | ||
[[nn:Deoksyribonukleinsyre]] |
[[nn:Deoksyribonukleinsyre]] |
||
[[no:DNA]] |
[[no:DNA]] |
||
[[nov:DNA]] |
|||
[[oc:Acid desoxiribonucleïc]] |
[[oc:Acid desoxiribonucleïc]] |
||
[[pam:DNA]] |
[[pam:DNA]] |
||
Version vom 17. Mai 2007, 14:14 Uhr
Die Desoxyribonukleinsäure (lat.-fr.-gr. Kunstwort) ist eine in allen Lebewesen vorkommende Nukleinsäure (lat. nucleus, Fruchtkern), die als Trägerin der Erbinformationen dient. Sie enthält die für die biologische Entwicklung und das Überleben der Zelle notwendige genetische Information. Im Normalzustand ist sie in Form einer Doppelhelix organisiert. Im deutschen Sprachgebrauch wird die Desoxyribonukleinsäure zunehmend mit der englischen Abkürzung DNA (deoxyribonucleic acid) bezeichnet, „veraltend“[1] auch noch mit DNS.
Die DNA ist ein langes Kettenmolekül (Polymer) aus Einzelstücken, sogenannten Nukleotiden. Jedes Nukleotid besteht aus einem Phosphat-Teil, dem Zucker Desoxyribose und einer von vier organischen Basen mit den Kürzeln A,T, G und C. Die Abfolge der Basen legt die Abfolge der Aminosäuren von Proteinen mit Hilfe des genetischen Codes fest, jeweils drei Basen stehen für eine Aminosäure. Bei den Zellen von Pflanzen, Tieren, Pilzen und vielen Einzellern, den sogenannten Eukaryoten ist der Großteil der DNA im Zellkern lokalisiert, während bei Bakterien und Archaeen (den Prokaryoten) die DNA im Zellinneren, dem Cytoplasma, verteilt vorliegt. Bestimmte Arten von Zellorganellen, die Mitochondrien und Chloroplasten, enthalten ebenfalls DNA. Lediglich bestimmten Viren, den sogenannten RNA-Viren fehlt die DNA, hier wird die genetische Information durch das ähnliche Molekül Ribonukleinsäure (RNA) kodiert.
Entdeckungsgeschichte


Entdeckt wurde die DNA bereits 1869 von dem Schweizer Arzt Friedrich Miescher in einem Extrakt aus Eiterzellen, der in Zellkernen das Nuklein vorfand, jedoch die Funktion dieser Substanz noch nicht sicher bestimmen konnte. Miescher arbeitete damals im Labor von Felix Hoppe-Seyler im Tübinger Schloss.[2] Erst 1929, 60 Jahre später, identifizierte Phoebus Levene die Bestandteile der DNA (Base, Zucker und Phosphatrest).[3] Levene schlug eine kettenartige Struktur der DNA vor, in welcher die Nukleotide durch die Phosphatreste zusammengefügt und sich stetig wiederholen. 1937 publizierte William Astbury erstmals Röntgenbeugungsmuster, welche auf eine repetitive Struktur der DNA hinwiesen.[4]
Oswald Theodore Avery entdeckte 1943, dass ein Stamm der inaktiven Form von Pneumococcus ihre Infektionsfähigkeit wiedergewinnen konnten, wenn sie mit einem abgetöteten Stamm der infektiösen Form zusammengebracht wurden. Avery identifiziert die DNA als die Substanz, welche die Information für diese Transformation beherbergte.[5] Unterstützung in seiner Interpretation erhielt Avery 1952, als Alfred Hershey und Martha Chase nachwiesen, dass die DNA das genetische Matrial das T2 Phagen ist.[6]
Die wirkliche Struktur der DNA wurde im Jahre 1953 jedoch von dem US-Amerikaner James Watson und dem Briten Francis Crick in ihrem berühmten Artikel Molecular structure of nucleic acids. A structure for deoxyribose nucleic acid aufgeklärt[7]. Watson kam 1951 nach England, nachdem er ein Jahr zuvor an der Indiana University in den USA promoviert hatte. Er hatte zwar ein Stipendium für Molekularbiologie bekommen, beschäftigte sich aber vermehrt mit der Frage des menschlichen Erbguts. Crick widmete sich gerade erfolglos seiner Promotion über die Kristallstruktur des Hämoglobinmoleküls in Cambridge, als er 1951 Watson traf. Zu dieser Zeit war bereits ein erbitterter Wettlauf um die Struktur der DNA entbrannt, an dem sich mehrere andere Laboratorien beteiligten (unter auch Linus Pauling am California Institute of Technology). Obwohl Watson und Crick eigentlich anderen Projekten zugeteilt waren und kein grosses Fachwissen in Chemie besassen, beteiligten sie sich an dem Wettlauf um die Strukturaufklärung und bauten ihre Überlegungen auf den Forschungsergebnissen der anderen Wissenschaftler auf. Watson besuchte Ende 1952 Maurice Wilkins am King's College in London, der ihm DNA-Röntgenaufnahmen von Rosalind Franklin zeigte (was gegen den Willen von Franklin geschah). Watson sah sofort, dass es sich bei dem Molekül um eine Doppel-Helix handeln musste; Franklin selber hatte aufgrund der Daten auch das Vorhandensein einer Helix vermutet, jedoch hatte sie kein überzeugendes Modell für die Struktur vorzuweisen. Da bekannt war, dass die Pyridin und Pyrimidin-Basen Paare bilden, gelang es Watson und Crick, die komplette Molekularstruktur herzuleiten. So entwickelten sie am Cavendish-Laboratorium der Cambridge University das Doppelhelix-Modell der DNA mit den Basenpaaren in der Mitte, welches am 25. April 1953 in der Zeitschrift Nature publiziert wurde.[8] Diese denkwürdige Publikation endete mit dem Satz "It has not escaped our notice that the specific pairity we have postulated immediately suggests a possible copying mechanism for the genetic material" (Es ist unserer Aufmerksamkeit nicht entgangen, dass die speziellen Paarungen, die wir als gegeben voraussetzen, unmittelbar auf einen möglichen Vervielfältigungsmechanismus für die genetische Erbsubstanz schließen lassen.)
„Für ihre Entdeckungen über die Molekularstruktur der Nukleinsäuren und ihre Bedeutung für die Informationsübertragung in lebender Substanz“ erhielten Watson und Crick zusammen mit Maurice Wilkins 1962 den Nobelpreis für Medizin.[9] Rosalind Franklin, deren Röntgenbeugungsdiagramme wesentlich zur Entschlüsselung der DNA-Struktur beigetragen hatten, war zum Zeitpunkt der Nobelpreisverleihung bereits verstorben und erhielt deshalb keinen Nobelpreis.
Aufbau der DNA


Die Desoxyribonukleinsäure ist ein langes Kettenmolekül (Polymer) aus vielen Einzelbausteinen, die man Desoxyribonukleotide nennt. Jedes Nukleotid ist eine Verbindung aus einem Phosphorsäure-Molekül, dem Zucker Desoxyribose sowie einer heterozyklischen Nukleobase: Adenin (A), Thymin (T), Guanin (G) oder Cytosin (C). Die Desoxyribose- und Phosphorsäure-Untereinheiten sind bei jedem Nukleotid gleich und werden auch als "Rückgrat" des Moleküls bezeichnet; die vier verschiedenen Nukleotide unterscheiden sich also nur durch ihre Base und entsprechend werden die Abkürzungen A, T, G und C auch für die entsprechenden Nukleotide verwendet.
Die fünf Kohlenstoffatome einer Desoxyribose sind von 1' (sprich Eins Strich) bis 5' nummeriert. Am 1'-Ende dieses Zuckers sitzt die Nukleobase und am 5'-Ende hängt der Phosphorsäure- oder Phosphatrest. Eigentlich handelt es sich bei der Desoxyribose um die 2-Desoxyribose, da die OH-Gruppe (Alkohol) an der Position 2' fehlt (bzw. durch einen Wasserstoff ersetzt wurde). An der Position 3' jedoch befindet sich immer noch eine OH-Gruppe, welche die Desoxirobose über eine Phosphatgruppe zum 5' Kohlenstoffatom des jeweils nächsten Zuckers verknüpft, eine sogenannte Phosphodiester Bindung (siehe Abbildung). Somit besitzt jedes DNA-Molekül zwei verschiedene Enden: Ein 5' Ende und ein 3' Ende. Neue Bausteine werden immer an die OH-Gruppe am 3' Ende angefügt (die Kette kann also nur an einem Ende verlängert werden, dies hat Implikationen in der DNA-Replikation), indem ein Nukleosidtriphosphate (mit drei Phosphatresten) als neuer Baustein angeliefert wird und zwei Phosphate in Form von Pyrophosphat entfernt werden und der verbleibende Phosphatrest des jeweils neu hinzukommenden Nukleotids mit der OH-Gruppe am 3'-Ende des letzten im Strang vorhandenen Nucleotids unter Wasserabspaltung reagiert. Auf diese Weise können zahlreiche weitere Nukleotide an das 3'-Ende eines DNA-Einzelstrangs angehängt werden.
Jede Desoxyribose hat an ihrer 1' Position eine Base gebunden (A, G, C oder G), deren Abfolge schlussendlich die genetische Information kodieren. Während sich die Desoxyribosen verbunden durch Phosphat wie die "Stränge" einer Strickleiter winden, stellen die Basen dabei die "Sprossen" der Leiter dar. Dabei werden immer zwei Basen gepaart, jeweils ein Purin (Adenin bzw. Guanin) mit einem Pyrimidin (Thymin bzw. Cytosin ). Zwischen Adenin und Thymin bilden sich dabei zwei Wasserstoffbrücken aus; Cytosin und Guanin sind über drei Wasserstoffbrücken miteinander verknüpft. Es bilden sich also in den "Sprossen" keine Atombindungen aus sondern lediglich schwache Wasserstoffbrücken, was es ermöglicht, die beiden Stränge auch wieder zu trennen. Dadurch, dass immer ein Purin mit einem Pyrimidin kombiniert wird, bleibt der Abstand zwischen den Strängen (also die Länge der "Sprossen") gleich und eine regelmässige Struktur kann entstehen.
Normalerweise ist DNA rechtshändig gedreht mit den oben genannten Strukturmerkmalen. Neben dieser, auch „B-DNA“ genannten Konformation, existieren auch eine „A-Form“ sowie eine 1979 von Alexander Rich und seinen Kollegen am MIT erstmals auch untersuchte, linkshändige, so genannte „Z-DNA“. Diese tritt besonders in G-C-reichen Abschnitten auf. Erst 2005 wurde über eine Kristallstruktur berichtet, welche Z-DNA direkt in einer Verbindung mit B-DNA zeigt und so Hinweise auf eine biologische Aktivität von Z-DNA liefert. [10]
 |
 |
Es existieren drei Strukturmodelle der DNA (die A, B und Z-DNA) wobei die biologisch wichtigste die "B-Form" ist, wie sie auch von Watson und Crick vorgeschlagen wurde. Hier wird nur auf die B-Form eingegangen, die Tabelle weiter unten zeigt die Unterschiede der drei Formen im direkten Vergleich.
Die DNA besteht aus zwei langen Molekülketten, die schraubenförmig um eine gemeinsame, gedachte Achse gewunden sind (Doppelhelix-Struktur). Die Helix besteht aus einem Rückgrat aus alternierenden Phosphatresten und Zuckerresten (2-Desoxribosen), an welche jeweils die zum Zentrum der Helix gerichteten Basen gebunden sind. Der Drehsinn der beiden Ketten (DNA-Einzelstränge) ist in der B-DNA jeweils rechts und die beiden Ketten sind antiparallel, sie besitzen also jeweils eine komplementäre Basensequenz. Die ganze Helix hat einen Durchmesser von ungefähr 2 nm und windet sich mit jedem Zuckermoleküle um 0.34 nm nach oben. Die Ebenen der Zuckermoleküle stehen in einem Winkel von 36° zueinander und eine vollständige Drehung wird folglich nach 10 Basen (360°) und 3.4 nm erreicht. Die Phosphatreste, welche den Rand der Helix besetzen und augrund ihrer negativen Ladung hydrophil sind, bescheren der DNA selbst eine negative Ladung. In der Mitte der Helix befinden sich die Purin- bzw. Pyrimidinbasen, welche über Wasserstoffbrücken miteinander wechselwirken und durch hydrophobe Wechselwirkungen die Helixstruktur stabilisieren. Damit die Helix einen einheitlichen Durchmesser behält, besteht ein Basenpaar immer aus einer Purin- und einer Pyrimidinbase (A paart mit T und G mit C). Die Wasserstoffbrücken werden zwischen den Molekülpositionen 1=1 und 6=6 aufgebaut. Bei Guanin und Cytosin, die im Gegensatz zum Basenpaar A-T drei Wasserstoffbrücken besitzen, bildet sich zusätzlich zwischen 2=2 eine H-Brücke aus.
Die Basenabfolge in einer Kette ist zwar beliebig, aber für ein bestimmtes DNA-Molekül meist charakteristisch, da es Information speichert (analog zu einem Buch, das auch mit nur wenigen Zeichen auskommt aber durch verschiedenartiges Aneinanderreihen der Buchstaben Information speichern kann - im Code der DNA gibt es aber nur 4 und nicht 26 "Buchstaben"). Obwohl ein einzelnes Basenpaar sehr klein ist, werden die DNA-Moleküle sehr lange, da Millionen Basenpaare aneinandergereiht werden können. So ist das grösste menschliche Chromosom (Chromosom Nr. 1) über 220 Millionen Basenpaare lang.[11]

| Strukturmerkmal | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| helikaler Drehsinn | rechts | rechts | links |
| Durchmesser | ~26 Å | ~20 Å | ~18 Å |
| Basenpaare pro helikale Windung | 11.6 | 10.0 | 12 (6 Dimere) |
| Helikale Windung je Basenpaar (twist) | 31° | 36° | 60° (pro Dimer) |
| Ganghöhe (Anstig pro Windung) | 34 Å | 34 Å | 44 Å |
| Anstieg pro Base | 2.9 Å | 3.4 Å | 7.4 Å (pro Dimer) |
| Neigungswinkel der Basenpaare zur Achse | 20° | 6° | 7° |
| Große Furche | eng und tief | breit und tief | flach |
| Kleine Furche | breit und flach | eng und tief | eng und tief |
| Zuckerkonformation | C3'-endo | C2'-endo | Pyrimidine: C2'-endo Purine: C3'-endo |
| Glykosidische Bindung | anti | anti | Pyrimidine: anti Purine: syn |
Der genetische Informationsgehalt der DNA
Bestimmte Abschnitte der DNA, die so genannten Gene, kodieren für genetische Informationen, die dem Organismus nützlich ist. Gene enthalten „Baupläne“ für Proteine oder Moleküle, welche bei der Proteinsynthese oder Regulation des Stoffwechsels einer Zelle beteiligt sind. Die Reihenfolge der Basen bestimmt dabei die genetische Information. Diese Basensequenz kann mittels Sequenzierung z.B. über die Sanger-Methode ermittelt werden.
Die Basenabfolge (Basensequenz) eines Genabschnitts der DNA wird zunächst durch die Transkription in die komplementäre Basensequenz eines so genannten Ribonukleinsäure-Moleküls überschrieben (abgekürzt RNA, selten auch deutsch RNS). RNA enthält im Unterschied zu DNA den Zucker Ribose anstelle von Desoxyribose und die Base Uracil anstelle von Thymin, der Informationsgehalt ist aber derselbe. Für die Proteinsynthese werden sogenannte mRNAs verwendet, einsträngige RNA-Moleküle, die aus dem Kern ins Cytoplasma hinaustransportiert werden, wo die Proteinsythese stattfindet (siehe Proteinbiosynthese).
Die sogenannte „Ein-Gen-Ein-Protein-Hypothese“, die besagt, dass von einem kodierenden Abschnitt auf der DNA die Sequenz eines Proteinmoleküls abgelesen wird, hat heute nur noch eingeschränkte Gültigkeit: Es gibt Regionen der DNA, die durch Verwendung unterschiedlicher Leseraster bei der Transkription jeweils mehrere Proteine kodieren. Ausserdem können durch alternierendes Splicing (nachträgliches Schneiden der mRNA) verschiedene Isoformen eines Proteins hergestellt werden.
Chromatin und Chromosomen
Organisiert ist die DNA in der eukaryotischen Zelle in Form von Chromatinfäden, genannt Chromosomen, die im Zellkern liegen. Ein einzelnes Chromosom enthält jeweils einen langen, kontinuierlichen DNA-Doppelstrang. Da ein solcher DNA-Faden mehrere Zentimeter lang sein kann, ein Zellkern aber nur wenige Mikrometer Durchmesser hat, muss die DNA zusätzlich komprimiert bzw. „gepackt“ werden. Dies geschieht bei Eukaryoten mit sogenannten Chromatinproteinen, von denen besonders die basischen Histone zu erwähnen sind. Sie bilden die Nukleosomen um die die DNA auf der niedrigsten Verpackungsebene herumgewickelt wird. Während der Kernteilung (Mitose) wird jedes Chromosom zu einer kompakten Form kondensiert. Dadurch werden sie lichtmikroskopisch bereits bei geringer Vergrößerung sichtbar.
Bakterielle und virale DNA
In prokaryotischen Zellen liegt die DNA nicht als linearer Faden mit einem Anfang und einem Ende vor, sondern als zirkuläres Molekül - das 5'-Ende ist also mit dem 3'-Ende des DNA-Stranges verbunden. Diese ringförmig geschlossenen DNA-Moleküle werden je nach Länge der Sequenz als Bakterienchromosom oder Plasmid bezeichnet. Sie befinden sich bei Bakterien auch nicht in einem Zellkern, sondern liegen frei im Plasma vor. Die Prokaryoten-DNA wird mit Hilfe von Enzymen (z.B. Topoisomerasen und Gyrasen) zu einfachen „Supercoils“ aufgewickelt, die man sich wie eine verdrehte Telefonschnur vorstellen kann. Indem die Helices noch um sich selbst gedreht werden, sinkt der Platzbedarf für die Erbinformation.
Viren enthalten je nach Typ als Erbinformation entweder DNA oder RNA. Sowohl bei den DNA-Viren als auch bei den RNA-Viren wird die Nukleinsäure von einer Protein-Hülle geschützt.
DNA-Replikation
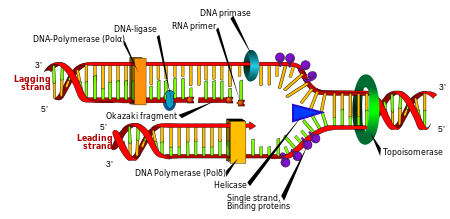

- siehe auch Hauptartikel Replikation
Die DNA ist in der Lage, sich mit Hilfe von Enzymen selbst zu verdoppeln. Sie wird nach dem so genannten semikonservativen Prinzip repliziert. Die doppelsträngige Helix wird zunächst durch das Enzym Helicase aufgetrennt. Jeder der beiden dabei entstehenden Einzelstränge dient nun als Matrize (Vorlage) für den jeweils zu synthetisierenden komplementären Gegenstrang, d. h. jedes der beiden Tochtermoleküle der replizierten DNA besteht aus einem alten und einem dazu komplementären, neu synthetisierten Einzelstrang.
Der Vorgang der DNA-Synthese, d. h. die Bindung der zu verknüpfenden Nukleotide, wird durch Enzyme aus der Gruppe der DNA-Polymerasen vollzogen. Ein zu verknüpfendes Nukleotid muss in der Triphosphat-Verbindung – also als Desoxyribonukleosidtriphosphat – vorliegen. Durch Abspaltung zweier Phosphatteile wird die für den Bindungsvorgang benötigte Energie frei.
Im Bereich der durch das Enzym Helicase gebildeten Replikationsgabel (das heißt, zweier auseinander laufender DNA-Einzelstränge) markiert zunächst ein RNA-Primer, der durch das Enzym Primase synthetisiert wird, den Startpunkt der DNA-Neusynthese. An dieses RNA-Molekül hängt die DNA-Polymerase dann ein zum Nukleotid des alten DNA-Einzelstrangs komplementäres Nukleotid, daran wieder ein weiteres neues passendes Nukleotid usw., bis die DNA wieder zu einem Doppelstrang komplettiert wurde. Dies geschieht an beiden geöffneten Einzelsträngen.
Dennoch entsteht dabei ein Problem: Die Verknüpfung der neuen Nukleotide zu einem komplementären DNA-Einzelstrang verläuft nur in 5'→3' Richtung, d. h. kontinuierlich den alten 3'→5'-Strang entlang (und dabei diesen ablesend) in Richtung der sich immer weiter öffnenden Replikationsgabel ohne Pause in einem Schritt durch.
Die Synthese des zweiten neuen Stranges am alten 5'→3'-Strang dagegen kann nicht kontinuierlich in Richtung der Replikationsgabel, sondern nur von dieser weg ebenfalls in 5'→3' Richtung erfolgen. Die Replikationsgabel ist aber zu Beginn der Replikation nur ein wenig geöffnet, weshalb an diesem Strang – in 'unpassender' Gegenrichtung – immer nur ein kurzes Stück neuer komplementärer DNA entstehen kann.
Da hier jeweils eine DNA-Polymerase nur ca. 1000 Nukleotide verknüpft, ist es notwendig, den gesamten komplementären Strang stückchenweise zu synthetisieren. Bei etwas weiter geöffnetem Zustand der Replikationsgabel lagert sich daher ein neuer RNA-Primer wieder direkt an der Gabelungsstelle an den DNA-Einzelstrang an, und die nächste DNA-Polymerase beginnt – sich von der Replikationsgabel entfernend – erneut ca. 1000 Nukleotide an den RNA-Primer zu hängen.
Derselbe Vorgang wird laufend wiederholt, d. h. der komplementäre DNA-Strang entsteht nach und nach häppchenweise. Bei der Synthese des 3'→5'-Stranges wird also pro DNA-Syntheseeinheit jeweils ein neuer RNA-Primer benötigt. Primer und zugehörige Syntheseeinheit bezeichnet man als Okazaki-Fragment. Die für den Replikations-Start benötigten RNA-Primer werden anschließend enzymatisch abgebaut. Dadurch entstehen Lücken im neuen DNA-Strang, welche durch spezielle DNA-Polymerasen mit DNA-Nukleotiden aufgefüllt werden.
Zum Abschluss verknüpft das Enzym Ligase die noch nicht miteinander verbundenen neuen DNA-Abschnitte zu einem einzigen, langen, komplementären Strang.
Nach Abschluss der Replikation wurden also zwei DNA-Einzelstränge in etwas unterschiedlicher Weise jeweils wieder zu einem Doppelstrang ergänzt. Aus einem DNA-Molekül sind somit zwei identische entstanden.
Andere Funktionen der DNA
DNA-Moleküle spielen als Informationsträger und „Andockstelle“ eine wichtige Rolle für Enzyme, die für die Transkription zuständig sind. Weiterhin ist die Information bestimmter DNA-Abschnitte, wie sie etwa in operativen Einheiten wie dem Operon vorliegt, wichtig für Regulationsprozesse innerhalb der Zelle.
Mutationen von DNA-Abschnitten – z. B. Austausch von Basen gegen andere oder Änderungen in der Basensequenz – führen zu Veränderungen des Erbgutes, die zum Teil tödlich (letal) für den betroffenen Organismus sein können. In seltenen Fällen sind solche Mutationen aber auch von Vorteil; sie bilden dann den Ausgangspunkt für die Veränderung von Lebewesen im Rahmen der Evolution. Mittels der Rekombination bei der geschlechtlichen Fortpflanzung wird diese Veränderung der DNA sogar zu einem entscheidenden Faktor bei der Evolution: Die eukaryotische Zelle besitzt in der Regel mehrere Chromosomensätze, d.h. ein DNA-Doppelstrang liegt mindestens zwei Mal vor. Durch wechselseitigen Austausch von Teilen dieser DNA-Stränge, dem Crossing-over bei der Meiose, können so neue Eigenschaften entstehen.
DNA-Schäden
DNA-Moleküle können durch verschiedene Einflüsse beschädigt werden. UV- oder γ-Strahlung, Alkylierung sowie Oxidation können die DNA-Basen chemisch verändern oder zum Strangbruch führen. Diese chemischen Änderungen beeinträchtigen unter Umständen die Paarungseigenschaften der betroffenen Basen. Dieses Prinzip ist eine wesentliche Ursache für Mutationen während der Replikation.
Einige häufige DNA-Schäden sind:
- die Bildung von Uracil aus Cytosin unter spontanem Verlust einer Aminogruppe durch Hydrolyse: Uracil ist wie Thymin komplementär zu Adenin.
- Thymin-Thymin-Dimerschäden (verursacht durch photochemische Reaktion zweier aufeinander folgender Thyminbasen im DNA-Strang durch UV-Strahlung, z.B. aus Sonnenlicht. Diese Schäden sind wahrscheinlich eine wesentliche Ursache für die Entstehung von Hautkrebs).
- die Entstehung von 8-oxo-Guanin durch Oxidation von Guanin: 8-oxo-Guanin ist sowohl zu Cytosin als auch zu Adenin komplementär. Während der Replikation können beide Basen gegenüber 8-oxo-Guanin eingebaut werden.
Aufgrund ihrer mutagenen Eigenschaften und ihres häufigen Auftretens (Schätzungen belaufen sich auf 104-106 neue Schäden pro Zelle und Tag) müssen DNA-Schäden rechtzeitig aus dem Genom entfernt werden. Zellen verfügen dafür über ein effizientes DNA-Reparatursystem. Dieses beseitigt Schäden mit Hilfe folgender Strategien:
- Direkte Schadensreversion: Ein Enzym macht die chemische Änderung an der DNA-Base rückgängig.
- Basenexcisionsreparatur: Die fehlerhafte Base, z. B. 8-oxo-Guanin, wird aus dem Genom ausgeschnitten. Die entstandene freie Stelle wird anhand der Information im Gegenstrang neu synthetisiert.
- Nukleotidexcisionsreparatur: Ein größerer Teilstrang, der den Schaden enthält, wird aus dem Genom ausgeschnitten. Dieser wird anhand der Information im Gegenstrang neu synthetisiert.
- Homologe Rekombination: Sind beide DNA-Stränge beschädigt, wird die genetische Information aus dem zweiten Chromosom des homologen Chromosomenpaars für die Reparatur verwendet.
- Replikation mit speziellen Polymerasen: DNA-Polymerase η kann z. B. fehlerfrei über einen TT-Dimerschaden replizieren. Menschen, bei denen Polymerase η nicht oder nur eingeschränkt funktioniert, leiden häufig an Xeroderma Pigmentosum, einer Erbkrankheit, die zu extremer Sonnenlichtempfindlichkeit führt.
Quellen
- ↑ laut Duden. Die deutsche Rechtschreibung. 22. Aufl., Mannheim, 2000
- ↑ Hubert Mania: Ein Opfer der wissenschaftlichen Vorurteile seiner Zeit. Die DNS wurde bereits 1869 im Tübinger Renaissanceschloss entdeckt. Telepolis. 17.04.2004.
- ↑ Levene P,: The structure of yeast nucleic acid. In: J Biol Chem. 40. Jahrgang, Nr. 2, 1919, S. 415 – 24 (jbc.org).
- ↑ Astbury W,: Nucleic acid. In: Symp. SOC. Exp. Bbl. 1. Jahrgang, Nr. 66, 1947.
- ↑ Avery O, MacLeod C, McCarty M: Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. In: J Exp Med. 79. Jahrgang, Nr. 2, 1944, S. 137 – 158 (jem.org).
- ↑ Hershey A, Chase M: Independent functions of viral protein and nucleic acid in growth of bacteriophage. In: J Gen Physiol. 36. Jahrgang, Nr. 1, 1952, PMID 12981234, S. 39 – 56 (jgp.org [PDF]).
- ↑ Watson, J.D. & Crick F.H. (1953): Molecular structure of nucleic acids. A structure for deoxyribose nucleic acid. In: Nature. Bd. 171, Nr. 4356, S. 737-738. PMID 13054692, http://www.nature.com/nature/dna50/watsoncrick.pdf
- ↑ Katharina Kramer: Dem Leben auf der Spur. GEO kompakt Nr. 7 (2006)
- ↑ Informationen der Nobelstiftung zur Preisverleihung
- ↑ Ha SC, Lowenhaupt K, Rich A, Kim YG, Kim KK: Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases. In: Nature. 437. Jahrgang, 2005, S. 1183–1186. PMID 16237447
- ↑ Gregory S, et al.: The DNA sequence and biological annotation of human chromosome 1. In: Nature. 441. Jahrgang, Nr. 7091, 2006, PMID 16710414, S. 315 – 21.
Literatur
- Chris R. Calladine u.a.: DNA - Das Molekül und seine Funktionsweise. Spektrum Akademischer Verlag, Heidelberg 2005 (3. Aufl.). ISBN 3-8274-1605-1
- Terence A. Brown: Moderne Genetik. Spektrum Akademischer Verlag, Heidelberg 1999 (2. Aufl.). ISBN 3827403065
- Ernst Peter Fischer: Am Anfang war die Doppelhelix. Ullstein, Berlin 2004. ISBN 3548366732
- Ernst Peter Fischer: Das Genom. Fischer, Frankfurt M 2002. ISBN 359615362X
- James D. Watson: Die Doppelhelix. Rowohlt, Reinbek 1997. ISBN 3499602555
- James D. Watson: Gene, Girls und Gamov. Piper, München 2003. ISBN 3-492-04428-X
- James D. Watson: Am Anfang war die Doppelhelix Ullstein, Berlin 2003. ISBN 3-550-07566-9
- James D. Watson, M. Gilman, J. Witkowski, M. Zoller: Rekombinierte DNA. Spektrum Akademischer Verlag, Heidelberg 1993 (2. Aufl.). ISBN 3860250728
- Thomas Lindahl: Instability and decay of the primary structure of DNA. in: Nature. Macmillan Jounals, London 1993,362, 709-715. ISSN 0028-0836
- W. Wayt Gibbs: Preziosen im DNA-Schrott. in: Spektrum der Wissenschaft. Heidelberg 2004,2, 68-75. ISSN 0170-2971
- W. Wayt Gibbs: DNA ist nicht alles. in: Spektrum der Wissenschaft. Heidelberg 2004,3, 68-75. ISSN 0170-2971
Weblinks
- Wiktionary: Desoxyribonukleinsäure – Bedeutungserklärungen, Wortherkunft, Synonyme, Übersetzungen
- Commons: Desoxyribonukleinsäure – Sammlung von Bildern, Videos und Audiodateien
- DNA / Aufbau / Graphik - Mutationen - Reparatur - Antibiotika - Zellbiologie - interzelluläre Genregulation
- DNA-Isolierung „in der Schulküche“
- DNA Interactive – Seite des Cold Spring Harbor Institute und des Howard Hughes Medical Institute (eine exzellente Einführung in die Thematik, engl.)
- DNA from the Beginning des Dolan DNA Learning Center
- „DNA from the Beginning“ (deutsch)
- Nukleinsäuren
- 3sat: Nano: Die größte biologische Entdeckung: 50 Jahre DNA-Struktur
- DNA – Aufbau und Vervielfältigung (Bestandteile & Aufbau der DNA, Replikation und PCR)




